El Presidente del Consejo de Seguridad Nuclear, el Rector de la Universidad Autónoma de Madrid, el Vicerrector de Investigación y de Transferencia de la Universidad Autónoma de Barcelona y la Rectora de la Universidad Rovira i Virgili han suscrito, con fecha 15 de noviembre de 2018, un Acuerdo para el estudio de la búsqueda de marcadores genéticos de sensibilidad a las bajas dosis de radiación en células linfoides humanas.
Para general conocimiento, y en cumplimiento de lo establecido en el artículo 48.8 de la Ley 40/2015, de 1 de octubre, de Régimen Jurídico del Sector Público, dispongo la publicación en el «Boletín Oficial del Estado» del referido Acuerdo, como anejo a la presente Resolución.
Madrid, 11 de diciembre de 2018.–El Presidente del Consejo de Seguridad Nuclear, Fernando Marti Scharfhausen.
REUNIDOS
De una parte: Don Fernando Marti Scharfhausen, Presidente del Consejo de Seguridad Nuclear (en adelante CSN), cargo para el que fue nombrado por el Real Decreto 1732/2012, de 28 de diciembre («BOE» número 313 del 29), en nombre y representación de este Organismo, con domicilio en la calle Justo Dorado, n.º 11, de Madrid y número de identificación fiscal Q2801036-A.
De otra parte: Don Rafael Garesse Alarcón, Rector de la Universidad Autónoma de Madrid (en adelante UAM), cargo por el que fue nombrado por Decreto 41/2013, de 20 de junio de 2017, del Consejo de Gobierno («BOCM» n.º 147, jueves 22) y cuyas competencias ostenta en virtud del artículo 20 de la Ley Orgánica 6/2001, de 21 de diciembre, de Universidades, parcialmente modificada por Ley Orgánica 4/2007, de 12 de abril, y los artículos 39 y 40 de los Estatutos de la UAM, aprobados por Decreto 214/2003, de 16 de octubre, modificados por Decreto 94/2009, de 5 de noviembre, con domicilio en la calle Einstein 3, Campus Universitario de Cantoblanco de Madrid, y número de identificación fiscal Q2818013A.
De otra parte: Don Armand Sánchez Bonastre, Vicerrector de Investigación y de Transferencia de la Universitat Autònoma de Barcelona (en adelante UAB), según nombramiento de 6 de junio de 2016, y haciendo uso de las atribuciones que le otorga la resolución de 15 de junio de 2016, que confiere autorización a los vicerrectores de Investigación y Transferencia e Innovación y de Proyectos Estratégicos, para la subscripción de convenios y contratos de Investigación por parte de la UAB, con sede social en el Campus Universitario s/n, 08193 Bellaterra (Cerdanyola del Vallès), y número de Identificación fiscal Q-0818002-H.
De otra parte: Doña María José Figueras Salvat, Rectora de la Universitat Rovira i Virgili (en adelante URV), cargo para el que fue nombrado por Decreto 32/2018, de 7 de junio («DOGC» 7638, del 8) que representa esta institución de acuerdo con las competencias que prevé el artículo 66 de Estatuto de la URV, aprobado por el Decreto 202/2003, de 26 de agosto («DOGC» 3963, de 8 de septiembre), y modificado por Acuerdo de Gobierno GOV/23/2012, de 27 de marzo («DOGC» 6100, de 2 de abril). La URV tiene el domicilio en la calle Escorxador s/n de Tarragona, y número de identificación fiscal Q-9350003-A.
Todas las Partes intervienen por sus respectivos cargos y en el ejercicio de las facultades que, para convenir en nombre de las Entidades a que representan, tienen conferidas y, a tal efecto,
EXPONEN
Que el CSN, como único organismo competente en materia de seguridad nuclear y protección radiológica, tiene legalmente asignada la función de evaluar el impacto radiológico de las instalaciones nucleares y radiactivas y de las actividades que impliquen el uso de radiaciones ionizantes, así como la de controlar y vigilar la calidad radiológica del medio ambiente de todo el territorio nacional.
Que el CSN suscribe el presente convenio en ejercicio de la función que le atribuye su Ley de Creación (Ley 15/1980, de 22 de abril) en su artículo 2, letra p), que es la de establecer y efectuar el seguimiento de planes de investigación en materia de seguridad nuclear y protección radiológica.
Que el CSN tiene suscrito un acuerdo marco de colaboración con la Universidad Autónoma de Madrid, de fecha 24 de julio de 1990, para la realización de actividades en el campo de la seguridad nuclear y la protección radiológica, siendo esta institución la que realizará la coordinación técnica de este proyecto a través del Centro de Biología Molecular Severo Ochoa.
Que en virtud del interés común de las Partes en la investigación acerca de los riesgos asociados a las bajas dosis de radiación, las Partes están interesadas en la búsqueda de marcadores genéticos de sensibilidad a las bajas dosis de radiación. La identificación de estos indicadores genéticos permitiría realizar una mejor estimación de los riesgos asociados a la salud en los trabajadores expuestos ocupacionalmente a las bajas dosis de radiación y a las personas sujetas a los procedimientos de radiodiagnóstico médico.
Que las Partes consideran que la UAM, a través del Departamento de Biología Celular e Inmunología del Centro de Biología Molecular Severo Ochoa (CBMSO), del que forma parte junto al Consejo Superior de Investigaciones Científicas (CSIC); que la UAB, a través del Departamento de Biología Celular, de Fisiología y de Immunología; y que la URV, a través del Laboratori de Toxicología y Salud Mediambiental (TecnATox), disponen de capacidad y experiencia suficientemente demostradas para llevar a cabo estudios como los descritos en el punto anterior, así como del personal necesario para realizarlo.
Que las Partes consideran que la colaboración entre ellas en este campo contribuirá al mejor cumplimiento de los objetivos propios de cada una de ellas, y aumentará el conocimiento científico y técnico en este ámbito en beneficio de todas las Partes.
Que el Acuerdo supone una cooperación entre CSN y las otras tres instituciones con la finalidad de garantizar que los servicios públicos que les incumben se prestan de modo que se logren los objetivos que tienen en común; y que el desarrollo de dicha cooperación se guía únicamente por consideraciones relacionadas con el interés público.
Por todo ello, las Partes convienen en formalizar el presente Acuerdo Específico con sujeción a las siguientes:
CLÁUSULAS
El objeto de este Acuerdo es la realización de un estudio encaminado a la búsqueda de marcadores genéticos de sensibilidad a las bajas dosis de radiación en células linfoides humanas con el objetivo de proporcionar nuevos instrumentos que permitan ser utilizados en los procesos de protección radiológica para valorar los riesgos en la población de la exposición a bajas dosis de radiación. El alcance de las actividades que se realizarán para alcanzar los objetivos expuestos se detalla en la Memoria Científica que se adjunta a este Acuerdo como Anexo A.
El presente Acuerdo entrará en vigor en la fecha de su firma, y tendrá una duración de tres años. No obstante, el Acuerdo podrá ser objeto de modificación o prórroga por mutuo acuerdo de las Partes, por un periodo máximo de otros cuatro años en el segundo caso, si fuera necesario rectificar las actividades acordadas o variar su plazo de ejecución. Para este caso, se formalizará la oportuna Cláusula Adicional con las condiciones de la prórroga o modificación.
Son obligaciones de la UAM, UAB y URV dentro de este Acuerdo Específico:
Realizar las actividades que se describen en la Memoria Científica que se adjunta como Anexo A, relacionadas con el objeto del Acuerdo descrito en la cláusula primera.
Poner a disposición del Acuerdo los recursos humanos necesarios para garantizar la máxima calidad de los trabajos en él incluidos.
Poner a disposición del CSN los métodos, metodologías, y, en general, toda la información y resultados que se generen durante la realización de las actividades objeto de este Acuerdo.
Documentar los trabajos realizados dentro del Acuerdo, en la forma que se describe en la Memoria Científica que se adjunta.
Son obligaciones del CSN dentro de este Acuerdo Específico:
Contribuir a la financiación del Acuerdo en la forma que se describe en la cláusula cuarta.
Poner a disposición de la UAM, UAB y la URV los datos u otra información que en su caso sea necesaria para la realización de los trabajos objeto de este Acuerdo.
Los costes asociados a las actividades incluidas en este Acuerdo se detallan en la Memoria Económica que se incluye como Anexo B. El coste total del Acuerdo comprenderá las partidas correspondientes a recursos humanos y materiales, así como las correspondientes a los viajes que se consideren necesarios y a los gastos de realización de análisis.
Con arreglo a las cantidades que figuran en dicha Memoria, los gastos correspondientes al proyecto, a lo largo de toda su duración, ascienden a un total de novecientos nueve mil cuatrocientos cincuenta y dos euros (909.452 €). El CSN contribuirá a la financiación del proyecto con la cantidad de trescientos veintiún mil trescientos ochenta y seis euros (321.386 €), cantidad en la que ya se han incluido todo otro tipo de gastos e impuestos. La diferencia entre lo aportado por el CSN y el presupuesto total del proyecto será aportada por otras fuentes de financiación, que se describen de forma pormenorizada en la Memoria Económica del proyecto que acompaña a este acuerdo.
La distribución de esta contribución del CSN se establece en aportaciones anuales a lo largo de tres ejercicios presupuestarios, correspondiendo a la aplicación presupuestaria con código 20.302.424M.640. Estas aportaciones anuales se abonarán mediante notas de pago emitidas por cada entidad participante (UAM, UAB y URV), en la forma y plazos que se detallan en la Memoria Económica, haciéndose los pagos en las correspondientes cuentas corrientes de cada una de las entidades que sean indicadas.
Las citadas cantidades serán satisfechas por el CSN previa entrega y aceptación de la documentación que se define en la Memoria Científica y en la Memoria Económica, y se abonarán condicionadas a la previa existencia de crédito específico y suficiente en cada ejercicio, con cumplimiento de los límites establecidos en el artículo 47 de la Ley General Presupuestaria.
Tanto el CSN como las otras cuatro instituciones realizan en el mercado abierto menos del 20% de las actividades objeto de la cooperación.
El CSN y las Partes designan respectivamente como responsables de la coordinación técnica del Acuerdo:
Por el CSN: Área de instalaciones radiactivas y exposiciones médicas de la Subdirección de Protección Radiológica Operacional del CSN.
Por la UAM: Javier Santos Hernández, Profesor Titular de Genética, que actuará como Coordinador de todo el Proyecto.
Por la UAB: Anna Genescá Garrigosa, Catedrática de Genética.
Por la URV: María Victoria Linares Vidal, Profesora Agregada de Fisiología.
Los coordinadores técnicos serán responsables de controlar el desarrollo de este Acuerdo específico, y de adoptar, por mutuo acuerdo, las decisiones necesarias para la buena marcha de las actividades contempladas en el mismo. Para ello, se reunirán con la frecuencia que consideren necesaria y podrán asesorarse de los expertos que consideren oportuno.
Las Partes conceden, con carácter general, la calificación de información confidencial a la obtenida en aplicación de este acuerdo de colaboración, por lo que asumen de buena fe el tratamiento de restricción en su utilización por sus respectivas organizaciones, a salvo de su uso para el destino o finalidad pactados.
En caso de que los trabajos o resultados del Acuerdo sean objeto de difusión, ya sea oral o escrita, por cualquiera de las Partes, para usos que no sean de carácter científico o académico, esta deberá ser aprobada por las Partes. Toda publicación, presentación o difusión por cualquier medio incluirá una mención explícita a la financiación parcial del estudio por parte del CSN.
Los resultados de las actividades que se realicen con la financiación proporcionada en este Acuerdo pertenecerán en exclusiva y por igual proporción a las Partes, así como a las otras entidades financiadoras cuando proceda.
Cualquiera de las Partes podrá rescindir o suspender temporalmente este Acuerdo, preavisando con al menos tres meses de antelación a la fecha en que la resolución deba ser efectiva. En tal caso, las Partes se comprometen a abonar el importe de los trabajos y gastos incurridos y los comprometidos a los que ineludiblemente haya que hacer frente pese a la resolución del Acuerdo.
En el supuesto de que la interrupción se produjera por incumplimiento culpable de alguna de las Partes, procederá restablecer previamente el equilibrio financiero del Acuerdo, en el estado en el que se encuentre. Toda Parte que no realice las gestiones necesarias para dicho restablecimiento, y en tanto en cuanto no se produzcan, quedará excluida de la parte de la propiedad en los resultados, así como su derecho de explotación para cualquier fin.
La UAM, UAB y URV entregarán al CSN un informe conjunto de los resultados parciales obtenidos hasta el momento de la interrupción y podrán utilizar libremente dichos resultados, siempre que se salvaguarden las condiciones estipuladas en las cláusulas Sexta y siguientes.
El presente Acuerdo, por su propia naturaleza, está excluido de la aplicación de la Ley 9/2017, de 8 de noviembre, de Contratos del Sector Público, sometiéndose a la Ley 40/2015, de 1 de octubre, de Régimen Jurídico del Sector Público, las demás normas generales de Derecho Administrativo, los principios de buena administración y al ordenamiento jurídico en general.
La interpretación del Acuerdo se realizará bajo el principio de buena fe y confianza legítima entre las Partes, que convienen en solventar de mutuo acuerdo las diferencias que pudieran presentarse en su aplicación. Para ello, surgida la controversia, cada Parte designará un representante si bien, en el caso de no lograrse común acuerdo, las Partes someterán la cuestión al conocimiento y competencia de la Jurisdicción Contencioso-Administrativa, de conformidad con lo establecido en la Ley 29/1998, de 13 de julio, reguladora de la Jurisdicción Contencioso-Administrativa.
Habiendo leído el presente por sí mismos y hallándose conformes, lo firman por cuadruplicado ejemplar y a un solo efecto en Madrid, a 15 de noviembre de 2018. Por el Consejo de Seguridad Nuclear, el Presidente, Fernando Marti Scharfhausen.–Por la Universidad Autónoma de Madrid, el Rector, Rafael Garesse Alarcón.–Por la Universidad Autónoma de Barcelona, el Vicerrector de Investigación y de Transferencia, Armand Sánchez Bonastre.–Por la Universidad Rovira i Virgili, la Rectora, María José Figueras Salvat.
Búsqueda de marcadores genéticos de sensibilidad a las bajas dosis de radiación en células linfoides humanas
Información de las instituciones participantes:
|
N.º participante |
Acrónimo |
Nombre de la institución participante |
|---|---|---|
|
1 |
UAM |
Universidad Autónoma de Madrid. |
|
2 |
UAB |
Universidad Autónoma de Barcelona. |
|
3 |
URV |
Universidad Rovira i Virgili. |
Coordinación del proyecto: Universidad Autónoma de Madrid, Centro de Biología Molecular Severo Ochoa. c/ Nicolás Cabrera, 1. 28049-Madrid.
Nombre del coordinador del proyecto: Prof. Javier Santos Hernández.
Teléfonos de contacto del coordinador: 91 497 82 00 / 91 196 46 27.
Dirección de correo electrónico: javier.santos@uam.es
Índice:
A.1 Resumen de la propuesta.
A.2 Finalidad del proyecto.
A.3 Antecedentes y estado actual del tema.
A.4 Hipótesis de trabajo y objetivos.
A.5 Plan de trabajo y metodologías asociadas.
A.6 Descripción de los grupos participantes.
A.7 Beneficios del proyecto, difusión y explotación de los resultados.
A.8 Contribución del proyecto al plan estratégico de I+D del CSN.
A.1 Resumen de la propuesta.
La radiación ionizante se considera un potente agente carcinogénico sin un umbral de dosis a partir del cual se aprecian sus efectos. Sin embargo, la cuantificación del riesgo de las exposiciones encontrado a bajas dosis de radiación sigue siendo problemática y está sujeta a incertidumbres. Por lo tanto, el gran desafío de la investigación en radiobiología consiste en proporcionar datos que contribuyan a mejorar el conocimiento de las bases genético-moleculares de la respuesta celular a las exposiciones con bajas dosis de radiación ionizante. Este proyecto pretende, por un lado identificar nuevos genes de sensibilidad a las bajas dosis de radiación, utilizando un modelo in vitro de células linfoides y apoyándose en un enfoque innovador basado en un análisis genético a gran escala que integre tanto los cambios en la expresión génica a nivel del ARN (transcriptoma) y las proteínas (proteoma) como las modificaciones en los patrones de metilación del ADN (epigenoma) y en los niveles de fosforilación de las proteínas (fosfoproteoma). La variabilidad individual en el riesgo de cáncer relacionado con la radiación y la susceptibilidad genética al cáncer son dos aspectos de gran relevancia que deben ser tenidos en cuenta en términos de protección radiológica. Por tanto, nuestra propuesta proyecto se plantea también validar los genes de respuesta a las bajas dosis de radiación identificados en el modelo in vitro en linfocitos de sangre periférica obtenidos de trabajadores expuestos ocupacionalmente a la radiación, con el fin de definir marcadores genéticos de radiosensibilidad individual que se puedan incorporar a los protocolos de protección radiológica actuales.
A.2 Finalidad del proyecto.
Aunque los riesgos para la salud de las exposiciones a altas dosis de radiación ionizante son bien conocidos y son tratados eficazmente por las medidas de protección radiológica actuales, hoy en día existen incertidumbres acerca de los efectos biológicos asociados a las bajas dosis de radiación ionizante. Para dar respuesta a estos interrogantes, la Comisión Europea (CE) y cinco de los Estados miembros de la Unión Europea (UE) que participan en el Programa EURATOM establecieron en 2008 un grupo de expertos de alto nivel (High Level Experts Group, HLEG) con el objetivo de identificar las necesidades de investigación y proponer una mejor la integración de los esfuerzos europeos en el campo de la protección a las bajas dosis de radiación. En Abril de 2009, el HLEG propone la creación de una plataforma europea dedicada a la investigación de los riesgos que tiene sobre la salud la exposición a las bajas dosis, conocida como MELODI (Multidisciplinary European LOw Dose Initiative). In 2010 MELODI, se registró como asociación con 15 socios fundadores, entre ellos la UAM y la URV. A finales de Octubre de 2014, el número de instituciones incorporadas a MELODI hasta 31, con la incorporación entre otras de la UAB. Una de las principales actividades de MELODI es el establecimiento y actualización de una Agenda Estratégica de Investigación (Strategic Research Agenda, SRA) a largo plazo (> 20 años) sobre los efectos de las bajas dosis de radiación con vistas a implementar medidas que mejoren los programas actuales de protección radiológica en Europa.
La SRA pretende también orientar las prioridades de los programas nacionales y europeos de investigación y la preparación de convocatorias competitivas a nivel europeo. En el futuro, la investigación europea en protección radiológica se organizará dentro de un Programa de Acción Conjunta cofinanciado (European Joint Program, EJP). El objetivo de la EJP es reunir a los organismos de financiación pertinentes de la UE y los Estados miembros para integrar la investigación europea y para administrar las convocatorias de propuestas de investigación en protección radiológica en nombre de la Comisión Europea. Esta actividad se basará en los programas estratégicos de investigación desarrollados por las cinco plataformas europeas de investigación en protección radiológica con el objetivo de establecer la interacción y las sinergias entre las diferentes áreas de especialización. Las cinco plataformas de protección radiológica son MELODI (radiobiología), ALLIANCE (radioecología), NERIS (gestión de emergencias), EURADOS (cuestiones de dosimetría) y una plataforma médica. Una herramienta operativa importante para el establecimiento de la EJP es el proyecto europeo OPERRA (Open Project for European Radiation Research Area), financiado por el VII programa marco de la UE (FP7).
El proyecto OPERRA ha financiado dos convocatorias de ayudas a la investigación con el fin de establecer grupos colaborativos de investigación en protección radiológica. Uno de los proyectos financiados en la primera convocatoria es SOPRANO (Systems Oriented Prediction of Radiation Risk) y en él participan, entre otros, la UAM y la URV. El proyecto SOPRANO es una actividad de investigación de 36 meses que reúne el primer equipo de investigación interdisciplinario en Europa para llevar a cabo una estrategia basada en la biología de sistemas aplicado al estudio de los efectos de las bajas dosis de radiación. SOPRANO tiene una naturaleza de estudio piloto que pretende explorar los tres componentes fundamentales de la biología de sistemas (la generación de datos, el análisis bioinformático y el desarrollo de modelos matemáticos predictivos) como el primer paso para probar la hipótesis de que esta estrategia se traducirá en un modelo predictivo capaz de determinar el riesgo individual a la exposición con bajas dosis de radiación. El proyecto que presentamos pretende contribuir a la identificación de nuevos genes de respuesta a las bajas dosis de radiación, utilizando un modelo in vitro de células linfoides, con vistas a incorporar información nueva al modelo predictivo que se está desarrollando en SOPRANO.
El informe de la SRA de MELODI para el año 2015 establece que la variabilidad individual en el riesgo de cáncer relacionado con la radiación y la susceptibilidad genética al cáncer son dos aspectos de gran relevancia que deben ser tenidos en cuenta en términos de protección radiológica. Por tanto, en nuestro proyecto se plantea también validar los genes de respuesta a las bajas dosis de radiación identificados en las células linfoides como indicadores de radiosensibilidad individual en linfocitos de sangre periférica obtenidos de trabajadores expuestos ocupacionalmente a la radiación. Si de estas investigaciones se dedujera que existen diferencias individuales en el comportamiento de estos marcadores genéticos, dichos resultados tendrían una gran transcendencia porque se demostraría una susceptibilidad genética individual a los efectos de las bajas dosis de radiación. La demostración de este hecho nos llevaría a considerar la incorporación de estos biomarcadores genéticos en los procedimientos actuales de protección radiológica.
A.3 Antecedentes y estado actual del tema.
A.3.1 La susceptibilidad individual a las bajas dosis de radiación es un problema no resuelto de la protección radiológica.
Los efectos biológicos inducidos por la exposición de las células de mamífero a la radiación ionizante (IR) están estrechamente relacionados con las dosis de radiación y las tasas de dosis. La evidencia epidemiológica indica que las exposiciones de 0,2 a 3 Gy aumento del riesgo de cáncer de manera proporcional a la dosis de radiación recibida [1]. Los riesgos para la salud derivados de la exposición a bajas dosis de radiación ionizante se estiman utilizando una modelo lineal sin umbral de la relación dosis-respuesta para extrapolar observaciones hechas en dosis altas [2]. Sin embargo, exposiciones por debajo de los 0,1 Gy pueden resultar en un escenario complejo de respuestas celulares que pueden ser de protección o supra-letales dependiendo de cuál haya sido la dosis recibida [3].
De los informes del Grupo de Expertos de Alto Nivel de la Unión Europea sobre la investigación de los riesgos a las bajas dosis de radiación [4], de la agenda transitoria de investigación de DoReMi [5] y de la agenda científica de MELODI (Multidisciplinary European Low Dose Initiative) [6] se desprende que la edad, sexo, la edad que un individuo tiene cuando recibe la exposición, el estilo de vida, otras exposiciones ambientales, y especialmente las diferencias en la constitución genética individual, son factores que pueden modificar el riesgo sobre la salud de las exposiciones a las bajas dosis de radiación.
A.3.2 Un análisis genético global integrado puede contribuir a mejorar el conocimiento de las bases genéticas que subyacen en la susceptibilidad individual a las bajas dosis de radiación.
El advenimiento de las nuevas tecnologías analíticas y computacionales ha dado lugar a una transformación rápida y fundamental en nuestra comprensión de la biología humana. Esto ha llevado al descubrimiento de los niveles de complejidad que rigen las respuestas biológicas, incluidos los que afectan a las radiaciones ionizantes. Los enfoques reduccionistas del pasado, que han asumido una relación de causalidad directa entre el daño del ADN y los resultados adversos que la falta de reparación o la reparación incorrecta de dicho daño tienen sobre la salud, resultan claramente insuficientes. Un estudio genético global que integre análisis no sólo del ADN sino también a nivel del ARN y de las proteínas, es un enfoque metodológico que en la investigación biomédica se puede utilizar para tener una imagen mucho más completa de un determinado proceso biológico.
El uso de enfoques basados en las técnicas ómicas ha contribuido a una mejor comprensión de la complejidad de las redes de regulación de las respuestas biológicas. El término «ómicas» agrupa a una serie de aproximaciones experimentales que tienen como objetivo llevar a cabo un análisis a nivel global de una muestra biológica. Entre ellas, la genómica se encarga del análisis del genoma completo de una célula a nivel del ADN. La transcriptómica, la proteómica y la metabolómica se las agrupa bajo la denominación de genómica funcional, ya que estudian a nivel global los productos de la expresión de los genes (ARN, proteínas y metabolitos). La proteómica no solo se limita a analizar el resultado de la expresión génica, sino que también estudia las modificaciones post-traduccionales que pueden sufrir las proteínas, así como la interacción entre ellas. Cuando se aborda desde un punto de vista global el estudio de las modificaciones epigenéticas analizando los patrones de metilación del ADN o las modificaciones postraduccionales de las histonas hablamos de la epigenómica. Todas las «ómicas» se basan en el análisis de un gran volumen de datos, y por lo tanto se valen de la bioinformática y de técnicas rápidas y automatizadas de alto rendimiento.
Durante la última década se han venido publicando numerosos estudios basados en el uso de líneas celulares humanas [7-13] y de modelos in vivo de ratón [14-15] que ponen de manifiesto diferencias notables en los perfiles de expresión génica a nivel del transcriptoma en las células irradiadas con bajas y altas dosis de radiación ionizante. Estos datos sugieren que los cambios en la expresión génica se pueden utilizar como un biomarcador de sensibilidad a la exposición con bajas dosis de radiación ionizante. Además, los análisis del transcriptoma en células obtenidas de machos y hembras de ratón tratados con bajas dosis de radiación ha demostrado la existencia de cambios significativos en la expresión génica global entre ambos sexos [16]. Por otro lado, un estudio a nivel del proteoma logró identificar una serie de proteínas que estaban fosforiladas en distintos tipos celulares expuestos a bajas dosis de radiación ionizante [17]. Sin embargo, el valor científico de los datos aportados por estos estudios ha sido escaso, ya que cada conjunto de datos se ha generado mediante un sistema modelo diferente con exposiciones a diferentes dosis y tiempos de análisis y donde el tipo celular variaba de unos experimentos a otros.
Un estudio reciente en el que se compararon el daño cromosómico producido por la radiación y la respuesta transcripcional a nivel global en cepas de ratón con diferencias genéticas en su sensibilidad al cáncer mamario, demostró que la respuesta a la exposición con bajas dosis de radiación varía dramáticamente con el fondo genético [18]. Este tipo de descubrimientos ponen de manifiesto la potencialidad que tienen los enfoques basados en la biología de sistemas en la búsqueda de los factores genéticos asociados a la sensibilidad individual a las bajas dosis de radiación.
A.3.3 Las células linfoides como modelo para la identificación de marcadores genéticos de radiosensibilidad individual en trabajadores ocupacionalmente expuestos a la radiación.
La radiosensibilidad de los tejidos del cuerpo humano, definida como probabilidad de respuesta celular a una dosis fija de radiación ionizante, depende de las células que los componen. Por regla general, las células más radiosensibles son aquellas que presentan un mayor índice de división celular, un periodo más largo de la fase mitótica y un menor grado de diferenciación celular, siendo el tejido linfoide uno de los más vulnerables al efecto carcinogénico de las radiaciones ionizantes [19].
El reglamento de protección sanitaria de los trabajadores de instalaciones radioactivas contra las radiaciones ionizantes (RD 783, 2001, Art 40) establece un protocolo de vigilancia sanitaria específico que además de aportar información sobre historial laboral y clínico del trabajador, obliga a la realización de un control biológico y estudios complementarios específicos [20]. Una de estos controles son los análisis de sangre en los que se valoran distintos parámetros hematológicos y bioquímicos.
El análisis de la frecuencia de micronúcleos (pequeños cuerpos extranucleares que resultan de roturas cromosómicas o retrasos en la segregación cromosómica durante la anafase) en linfocitos de sangre de periférica es un indicador utilizado comúnmente en protección radiológica para medir los efectos de la exposición ocupacional a bajas dosis de radiación ionizante [21]. Uno de los principales inconvenientes del ensayo de micronúcleos (MN) en linfocitos humanos es su reducida sensibilidad para la detección de daño inducido por bajas dosis de radiación, debido a la alta variabilidad individual de las frecuencias de MN espontáneos. Este problema se consiguió resolver estudiando aquellos MN que contienen fragmentos acéntricos, los cuales surgen específicamente como consecuencia de la exposición a la radiación [22]. La introducción de procedimientos semi-automatizados en el análisis de fragmentos acéntricos asociados a los MN abre la posibilidad de utilizar este tipo de ensayo en análisis a gran escala de individuos ocupacionalmente expuestos a la radiación [23]. No obstante, este tipo de análisis tiene una limitación fundamental relacionada con las exposiciones prolongadas a las bajas dosis de radiación (dosimetría retrospectiva). Según algunos estudios, el análisis de MN en individuos expuestos ocupacionalmente a la radiación, dentro del rango de los 0,25-62,9 mSv acumulados durante periodos que van de 1 a 30 años, no permite demostrar diferencias significativas en la frecuencia de MN con la situación control [24]. La razón de esta falta de significación hay que buscarla en el hecho de que los MN representan aberraciones cromosómicas inestables con una persistencia limitada en condiciones in vivo [21]. Por tanto, la búsqueda de marcadores estables de radiosensibilidad resulta esencial para su utilización como biosimetros en la estimación del riesgo de la exposición ocupacional a la radiación ionizante.
El análisis del tanscriptoma en células de cerebro de ratones expuestos a bajas dosis de radiación ionizante ha demostrado que los perfiles de expresión génica de dichas células sufren variaciones a lo largo de tiempo, identificando genes de respuesta temprana transitoria (30 minutos post-irradiación) genes de respuesta más tardía (4 horas post-irradiación) y genes de respuesta temprana cuyos niveles de expresión persisten en el tiempo [14]. Resultados similares se han obtenido analizando una línea celular humana derivada de células madre mesenquimales, distinguiéndose genes de respuesta temprana (1-4 horas) y genes de respuesta tardía (12-24 horas) [13]. En este caso, además se comprobó que los genes de respuesta temprana sufrían variaciones significativas en sus niveles de expresión después de diferentes exposiciones, todas ellas dentro del rango de las bajas dosis. Esta dependencia de dosis de los genes de respuesta temprana se ha demostrado también en células linfoblastoides humanas [11-12]. Este tipo de evidencias ponen de manifiesto la utilidad que tienen los marcadores genéticos para estimar los efectos producidos por las exposiciones a bajas dosis de radiación. Sin embargo, no existen hasta la fecha ningún estudio en el que que se haya planteado la identificación de marcadores genéticos de radiosensibilidad en células linfoides a nivel de todo el genoma, validación como biodosímetros para estimar el riesgo de la exposición ocupacional a la radiación ionizante.
A.3.4 Referencias.
1. Brenner DJ, Mossman KL: Do radiation doses below 1 cGy increase cancer risks? Radiat Res 2005, 163: 692-693.
2. Kathren RL Pathway to a paradigm: the linear nonthreshold dose-response model in historical context. The American Academy of Health Physics 1995 Radiology Centennial Hartman Oration. Health Phys 1996, 70: 621-635.
3. Mullenders L, Atkinson M, Paretzke H, Sabatier L, Bouffler S: Assessing cancer risks of low-dose radiation. Nat Rev Cancer 2009, 9: 596-604.
4. High Level and Expert Group: Report on European Low Dose Risk Research. Office for Official Publications of the European Communities, 2009, pp 1-38.
5. DoReMi Network of Excellence: Low Dose Research towards Multidisciplinary Integration http://www.doremi-noe.net/
6. MELODI (Multidisciplinary European Low Dose Initiative: Strategic Research Agenda 2014. http://www.melodi-online.eu/
7. Amundson SA, Bittner M, Chen Y, Trent J, Meltzer P, Fornace AJ Jr: Fluorescent cDNA microarray hybridization reveals complexity and heterogeneity of cellular genotoxic stress responses. Oncogene 1999, 18: 3666-3672.
8. Franco N, Lamartine J, Frouin V, Le Minter P, Petat C, Leplat JJ, Libert F, Gidrol X, Martin MT: Low-dose exposure to gamma rays induces specific gene regulations in normal human keratinocytes. Radiat Res 2005, 163: 623-635.
9. Fachin AL, Mello SS, Sandrin-Garcia P, Junta CM, Donadi EA, Passos GA, Sakamoto-Hojo ET: Gene expression profiles in human lymphocytes irradiated in vitro with low doses of gamma rays. Radiat Res 2007, 168: 650-665.
10. Lanza V, Pretazzoli V, Olivieri G, Pascarella G, Panconesi A, Negri R: Transcriptional response of human umbilical vein endothelial cells to low doses of ionizing radiation. J Radiat Res 2005, 46: 265-276.
11. Wyrobek AJ, Manohar CF, Krishnan VV, Nelson DO, Furtado MR, Bhattacharya MS, Marchetti F, Coleman MA: Low dose radiation response curves, networks and pathways in human lymphoblastoid cells exposed from 1 to 10cGy of acute gamma radiation. Mutat Res 2011, 722: 119-130.
12. Long XH, Zhao ZQ, He XP, Wang HP, Xu QZ, An J, Bai B, Sui JL, Zhou PK: Dose-dependent expression changes of early response genes to ionizing radiation in human lymphoblastoid cells. Int J Mol Med 2007, 19: 607-615.
13. Jin YW, Na YJ, Lee YJ, An S, Lee JE, Jung M, Kim H, Nam SY, Kim CS, Yang KH, Kim SU, Kim WK, Park WY, Yoo KY, Kim CS, Kim JH: Comprehensive analysis of time- and dose-dependent patterns of gene expression in a human mesenchymal stem cell line exposed to low-dose ionizing radiation. Oncol Rep 2008, 19: 135-144.
14. Yin E, Nelson DO, Coleman MA, Peterson LE, Wyrobek AJ: Gene expression changes in mouse brain after exposure to low-dose ionizing radiation. Int J Radiat Biol 2003, 79: 759-775.
15. Lowe XR, Bhattacharya S, Marchetti F, Wyrobek AJ: Early brain response to low-dose radiation exposure involves molecular networks and pathways associated with cognitive functions, advanced aging and Alzheimer’s disease. Radiat Res 2009, 171: 53-65.
16. Kovalchuk O, Ponton A, Filkowski J, Kovalchuk I: Dissimilar genome response to acute and chronic low-dose radiation in male and female mice. Mutat Res 2004, 550: 59-72.
17. Marchetti F, Coleman MA, Jones IM, Wyrobek AJ: Candidate protein biodosimeters of human exposure to ionizing radiation. Int J Radiat Biol 2006, 82: 605-639.
18. Snijders AM, Marchetti F, Bhatnagar S, Duru N, Han J, Hu Z, Mao JH, Gray JW, Wyrobek AJ: Genetic differences in transcript responses to low-dose ionizing radiation identify tissue functions associated with breast cancer susceptibility. PLoS One 2012, 7:e45394.
19. UNSCEAR 2006 Report: Effects of ionizing radiation. Vol II, 2006, pp 122-123, United Nations Publications.
20. García Escandón F, Fernández González MA, Castell Salvá R, Valls Fontanals A. Radiaciones ionizantes. Protocolos de Vigilancia Sanitaria, 2003, Ministerio de Sanidad y Consumo.
21. Vral A, Fenech M, Thierens H: The micronucleus assay as a biological dosimeter of in vivo ionizing radiation exposure. Mutagenesis 2011, 26: 11-17.
22. Vral A, Thierens H, De Ridder L: In vitro micronucleus-centromere assay to detect radiation-damage induced by low doses in human lymphocytes. Int J Radiat Biol 1997, 71: 61-68.
23. Baeyens A, Swanson R, Herd O, Ainsbury E, Mabhengu T, Willem P, Thierens H, Slabbert JP, Vral A: A semi-automated micronucleus-centromere assay to assess low-dose radiation exposure in human lymphocytes. Int J Radiat Biol 2011, 87: 923-931.
24. Joseph LJ, Patwardhan UN, Samuel AM: Frequency of micronuclei in peripheral blood lymphocytes from subjects occupationally exposed to low levels of ionizing radiation. Mutat Res 2004, 564: 83-88.
A.4 Hipótesis de trabajo y objetivos.
La constitución genética juega un papel muy importante en la respuesta diferencial de los individuos frente a la exposición con bajas dosis de radiación ionizante. Por tanto, la identificación de marcadores genéticos que nos permitan determinar la radiosensibilidad individual en trabajadores expuestos ocupacionalmente a bajas dosis de radiación ionizante es de gran relevancia para la prevención y protección contra los efectos nocivos de la radiación sobre la salud, ya que la sensibilidad de las células a la radiación está íntimamente relacionada con la predisposición al cáncer. Los factores genéticos que modulan en cada individuo el riesgo sobre la salud de las exposiciones a bajas dosis de radiación ionizante se pueden identificar de manera efectiva mediante un enfoque experimental integrado basado en el análisis global del genoma a nivel del ADN, ARN y proteínas.
Para demostrar esta hipótesis de trabajo se han planteado los siguientes objetivos:
1. Identificar genes implicados en radiosensibilidad individual a partir de un análisis in vitro integrado que estudie de manera global los cambios en la expresión génica a nivel del transcriptoma y del proteoma, así como las alteraciones en los patrones de metilación del ADN (epigenoma) en una línea celular de origen linfoide expuesta a bajas dosis de radiación ionizante.
2. Validar genes de radiosensibilidad individual mediante un análisis in vivo de linfocitos de sangre periférica obtenidos a partir de una muestra de trabajadores sanitarios expuestos ocupacionalmente a bajas dosis de radiación ionizante, estratificada por sexo, edad y dosis recibida, para su utilización posterior como biomarcadores genéticos en los procedimientos de protección radiológica.
A.5 Plan de trabajo y metodologías asociadas.
A.5.1 Estrategia general del plan de trabajo.
Nuestra estrategia se centrará en un análisis genético a gran escala utilizando distintas tecnologías ómicas y las herramientas bioinformáticas necesarias para identificar genes que respondan diferencialmente a las bajas dosis de radiación ionizante en una línea celular linfoblastoide (estudio in vitro) y su validación en linfocitos de sangre periférica obtenida de trabajadores sanitarios implicados en procedimientos de diagnóstico o tratamiento médico con radiaciones para definir marcadores de sensibilidad relevantes para la protección radiológica (estudio in vivo).
Hemos seleccionado como modelo la célula linfoide, ya que combina un fenotipo sensible a las bajas dosis de radiación ionizante con la disponibilidad de líneas celulares linfoblastoides que derivan de linfocitos primarios de individuos sanos. Además, el tejido hematopoyético, a través de los análisis de sangre, tiene un papel importante en los controles sanitarios preceptivos que se hacen a los trabajadores ocupacionalmente expuestos a la radiación.
Las células linfoides serán tratadas con rayos X a una dosis de 0,02 Gy, que es equivalente al límite máximo de exposición permitido a los trabajadores ocupacionalmente expuestos a la radiación (20 mSv/año). Paralelamente, se irradiarán las células con una dosis alta de radiación de 2 Gy para determinar que genes se alteran exclusivamente en respuesta a las bajas dosis de radiación ionizante.
A.5.2 Desarrollo del plan de trabajo.
El presente proyecto se desarrollará en tres fases, cada una de ellas con una duración de un año. El plan de trabajo se ha organizado en base a cuatro tareas principales. La tarea 1 será la fase de generación de los datos genéticos a nivel global en el tipo celular seleccionado. Las células linfoblastoides irradiadas se analizarán a las 4 h después de finalizados los tratamientos, estudiando a nivel global los cambios en la expresión génica (transcriptoma y proteoma) y en los patrones de fosforilación de las proteínas (fosfoproteoma) y de metilación del ADN (metiloma) en respuesta temprana a la radiación. La tarea 2 se centrará en el análisis bioinformático de los datos generados por las distintas tecnologías ómicas, para construir un mapa molecular de la respuesta génica a las bajas dosis de radiación a distintos niveles de funcionalidad (transcriptoma, proteoma y epigenoma). Este mapa nos permitirá identificar genes de respuesta temprana y tardía a las bajas dosis de radiación en células linfoides. En la tarea 3 se llevará a cabo una validación de los genes de radiosensibilidad identificados, seleccionando aquellos que sean más relevantes desde el punto de vista de la respuesta a la radiación, en linfocitos de sangre periférica obtenidos de trabajadores sanitarios expuestos ocupacionalmente a unas bajas dosis de radiación, estudiando cambios de expresión (ARN y proteínas), alteraciones epigenéticas y modificaciones postraduccionales. Sobre estos genes se llevará un análisis de sus secuencias nucleotídicas para identificar posibles polimorfismos funcionales. Todos estos datos nos permitirán definir, a nivel genético, biomarcadores de sensibilidad individual a la radiación.
A.5.3 Representación esquemática de las tareas del plan de trabajo.
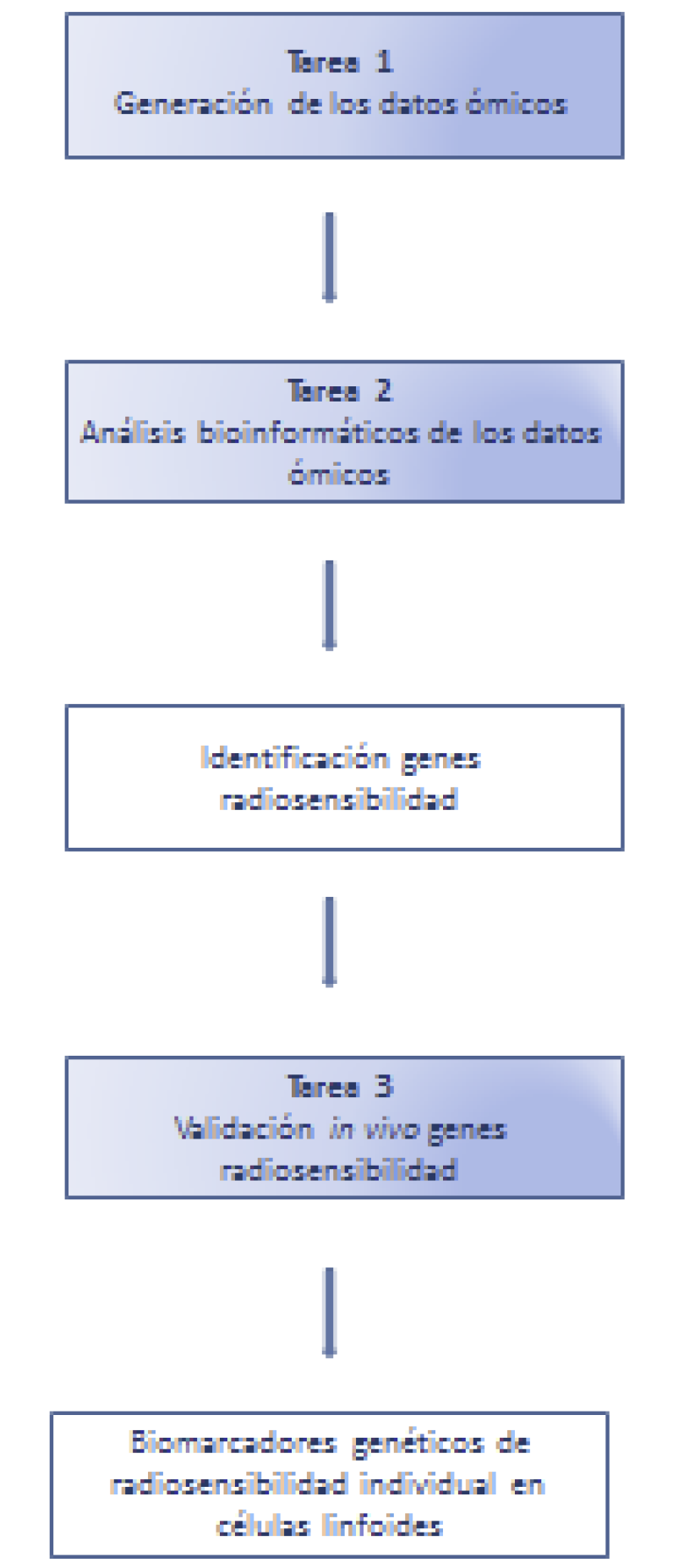
A.5.4 Descripción metodológica de las tareas del plan de trabajo.
A.5.4.1 Tarea 1. Generación de los datos ómicos obtenidos de células linfoblastoides humanas GM14511 expuestas a dosis bajas de rayos X.
a) Sub-tarea 1.1. Irradiación de células linfoides con 0,1 y 1 Gy de rayos X (UAM)
Se utilizará un único vial de células linfoides humanas CD3+ Pan T, derivadas de muestra de sangre periférica de una persona sana. Estas células serán irradiadas usando 100 kV de rayos X administrados en una dosis única de 0,1 Gy o 1 Gy. Paralelamente se utilizarán células no irradiadas como control. Las distintas condiciones experimentales se analizarán a las 6h post-irradiación para evaluar si existen diferencias genéticas en la respuesta temprana a las bajas dosis. Prepararemos tres replicados para cada dosis a partir de células con el mismo número de pases y procedentes del mismo stock pero crecidas en diferentes días. Una vez irradiadas, las células se recolectarán y se congelarán sus pellets. Para llevar a cabo los análisis ómicos correspondientes.
b) Sub-tarea 1.2. Análisis del transcriptoma (UAB).
Tras la exposición a radiación ionizante, las células modulan la expresión de genes asociados con las respuestas al daño en el ADN (DDR, DNA Damage Responses) con la finalidad de prevenir la propagación de mutaciones que pudieran originar inestabilidad genómica y cáncer. Las vías de respuesta al daño en el ADN dependen de la activación de una serie de genes cuyos productos coordinan la parada transitoria del ciclo celular, la remodelación de la cromatina y la reparación de la lesión o, en su defecto, la inducción de los procesos de apoptosis o senescencia. Si bien la mayor parte de los factores identificados hasta el momento son proteínas, estudios recientes indican la participación de RNAs largos no codificantes (lncRNA) en los procesos de DDR. Por tanto, la identificación de nuevos lncRNA que junto a los RNAs que codifican proteínas están implicados en las respuestas celulares al daño inducido en el ADN es importante para comprender la singularidad de los efectos de las bajas dosis de radiación.
El análisis del transcriptoma de las células mediante microarrays o micromatrices proporcionará datos sobre la expresión de decenas de millares de genes codificantes (mRNA) y no codificantes (lncRNA) regulados por exposición a dosis bajas de radiación. Para aplicar esta metodología de análisis global de la expresión génica se purifica el RNA total de las distintas muestras mediante tratamiento con Trizol y DNasa y se trabaja con triplicados técnicos de cada muestra para incrementar la robustez de los resultados. Para identificar los genes codificantes y no codificantes cuya expresión está regulada por bajas dosis de radiación se hibrida el RNA total de cada muestra (previamente retrotranscrito a cDNA) en una micromatriz ArrayStar Human LncRNA Expresión v3.0 que lleva unidos un total de 58.944 sondas de genes que codifican mRNA (26.109) y lncRNA (30.586). La micromatriz se utiliza para monitorizar de manera simultánea los niveles de expresión de miles de genes codificantes y no codificantes. Su funcionamiento consiste, básicamente, en medir el nivel de hibridación entre la sonda específica y la sonda de RNA problema pasado a cDNA, proporcionando para cada gen un nivel de fluorescencia emitida que es proporcional a su expresión. Dado que el RNA total de las muestras irradiadas y no irradiadas habrá sido marcado con fluoróforos distintos, la visualización de la matriz permitirá detectar los genes que se activan o reprimen en distintas condiciones de irradiación. La comparación de los niveles de cada uno de los RNA proporcionará datos relevantes acerca de los genes regulados por las bajas dosis de radiación.
c) Sub-tarea 1.3. Análisis epigenómico de los patrones de metilación del ADN (UAM).
La metilación en el carbono 5 de residuos de citosinas (5mC), presentes en las islas CpG de las regiones reguladoras de los genes, es un mecanismo epigenético usado por las células para controlar la expresión génica. Los cambios epigenéticos inducidos por la exposición a las bajas dosis de radiación ionizante incluyen tanto variaciones en el nivel global de metilación del ADN del genoma como alteraciones en la metilación del ADN en las regiones promotoras de genes específicos. Recientemente, se ha demostrado que la 5-hidroximetil citosina (5hmC), un producto de la desmetilación de la 5mC, juega un papel importante en la expresión transcripcional de los genes. Por tanto, en este trabajo analizaremos los niveles de 5-metil citosina (5mC) y 5-hidroximetil citosina (5hmC) a nivel genómico para definir los patrones específicos de metilación del ADN en respuesta a las bajas dosis de radiación.
Estudiaremos los niveles de 5mC (o 5hmC) en las regiones promotoras de genes específicos utilizando la micromatriz «Infinium MethylationEPIC BeadChip (Illumina) (Illumina Inc., San Diego, CA, USA). Esta micromatriz incluye las citosinas presentes en las regiones promotoras, en 5’UTR, en el primer exón, en el interior de la secuencia codificante y en 3’UTR, con el fin de proporcionar la visión más amplia y más completa del posible estado de metilación del ADN a nivel genómico. La micromatriz de Illumina detecta la metilación de citosinas (5mC) en las islas CpG en base al genotipado del ADN genómico modificado mediante un tratamiento con bisulfito (gDNA). Dicho tratamiento se llevará a cabo utilizando el «EZ DNA Methylation-Gold kit» (Zymo Research Corp., Irvine, CA, USA). Tras el tratamiento con bisulfito, las citosinas no metiladas se convierten en uracilo, mientras que las citosinas no metiladas se mantienen sin cambios. El ensayo interroga los diferentes genes químicamente diferenciados utilizando dos sondas específicas de sitio, una diseñada para el locus metilado y la otra para el locus no metilado. Estas sondas se utilizan como cebadores para una síntesis de ADN en donde se incorpora un ddNTP marcado, que posteriormente se tiñe con un reactivo de fluorescencia. La micromatriz de Illumina la utilizaremos también para cuantificar los niveles reales de 5mC y 5hmC. En este caso utilizaremos el «True Methyl Array kit» (Cambridge Epigenetics Ltd., Cambridge, UK) que permite la oxidación selectiva de la 5hmC a 5-formil citosina (5fC). En presencia de bisulfito, la 5fC y las citosinas no metiladas se transforman en uracilo. Un análisis comparativo de los resultados obtenidos de la plataforma Illumina a partir de muestras de ADN tratadas independientemente con los dos kits mencionados nos permitirá determinar los niveles reales de 5mC y 5hmC.
El estudio epigenómico se completará con el análisis de las modificaciones de histonas a nivel de dos residuos de la histona 3, lisina 4 trimetilada (H3K4me3, marca de activación génica) y lisina 9 monometilada (H3K9me1, marca inhibidora).
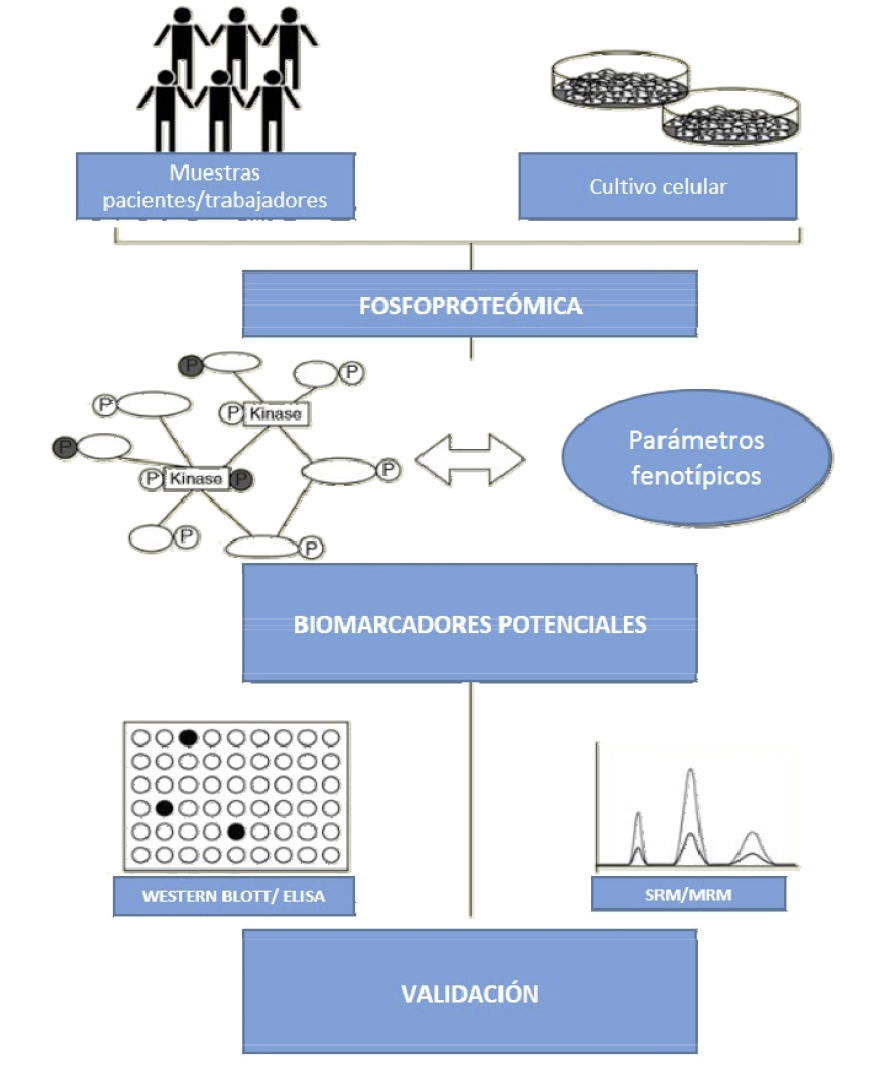
d) Sub-tarea 1.4. Análisis del proteoma y fosfoproteoma (URV).
Se procederá a la extracción de las proteínas y a una digestión proteolítica mediante un procedimiento de transferencia de fase surfactante. Los pellets de las células se homogenizaran mediante sonicación en un tampón de lisis y se determinará la concentración de proteínas mediante DC protein assay kit (Bio-Rad). Posteriormente se toma una muestra de 2mg del extracto de proteínas para iTRAQ o 500ug para SRM se someten a diferentes solventes y ácidos hasta conseguir recoger la fase acuosa que contiene los péptidos.
El enriquecimiento de fosfopéptidos se llevará a cabo mediante cromatografía de afinidad (Fe-IMAC). Los fosfopéptidos enriquecidos se marcan con iTRAQ4 plex (Applied Biosystems), siendo una de ellas la de referencia. Estos experimentos nos servirán para comparar los perfiles de fosforilación. Por otro lado, los péptidos marcados se fraccionan mediante HPLC mediante una columna e SCX (Agilent Technology). Los péptidos fraccionados se analizan mediante LTQ-Orbitrap y LC-SRM y se cuantifican.
e) Hitos de la tarea 1.
– H1.1: Compra de las células CD3+ Pan T y cultivo de las mismas en fase de pre-inmortalización (Mes 2) (UAM).
– H1.2: Finalización de la irradiación de las células y recolección de las mismas (Mes 4) (UAM).
– H1.3: Finalización del envío de todas las muestras (Mes 6) (UAM).
– H1.4: Obtención de todos los datos ómicos (Mes 18) (UAB, UAM, URV).
A.5.4.2 Tarea 2. Análisis bioinformático/bioestadístico de los conjuntos de datos ómicos obtenidos de células linfoides humanas CD3+ Pan T expuestas a dosis bajas de rayos X
a) Sub-tarea 2.1. Almacenaje de los datos ómicos (UAM).
Los datos ómicos generados en las tareas anteriores se almacenarán en los servidores compartidos del grupo UAM y ISCIII. La transferencia de archivos entre cliente/servidor se realiza siguiendo protocolos sftp que encriptan la sesión de conexión. Además cuenta con un sistema de control de usuario mediante contraseña y un control de IP (la dirección IP desde donde se produce la conexión para la transferencia de archivos debe facilitarse previamente a nuestro administrador, haciendo imposible la conexión desde otro lugar que no sea el indicado). Por último, el espacio disponible por defecto que se habilita para un almacenamiento nuevo de datos es de 2TB (pudiéndose ampliar si fuera necesario) y el software de conexión que se emplea bajo un sistema operativo Windows es el WINSCP o el FileZilla, ambos gratuitos. Si fuera necesario, se ha desarrollado un manual de conexión a los servidores que se puede facilitar para entrenar este proceso.
b) Sub-tarea 2.2. Análisis bioinformático/bioestadístico de los conjuntos de datos ómicos (UAM).
Primero se realizará un control de calidad de los datos que implicará la revisión de las muestras analizadas, valores obtenidos en los distintos análisis del transcriptoma, epigenoma y proteoma, y se valorará la imputación, recodificación y selección de las distintas variables recogidas.
En segundo lugar, utilizando herramientas bioinformáticas (librerías «annotate», «GO.db» de Bioconductor) se llevará a cabo la anotación de los distintos tipos de datos, así como la creación de una base de datos integrada de todo el proyecto.
En tercer lugar, se llevarán a cabo los análisis bioestadísticos para identificar las marcas genéticas a nivel del ARN, metilación y proteínas individualmente que se ven moduladas en respuesta a las bajas dosis de radiación ionizante en las células GM14511. Para ello se emplearán, entre otras, las funciones recogidas en las librerías «Limma», «topGO.db», «RnBeads», «methyAnalysis» y «methylkit» del proyecto R/Bioconductor. Se incluirán, además, análisis utilizando métodos de «Gene Set Enrichment» (GSEA) (GSEAPreranked).
c) Sub-tarea 2.3. Análisis integrativos (UAM).
Se llevará a cabo un análisis integrativo del proteoma, transcriptoma y metiloma para identificar las marcas genéticas, que nivel del ARN, metilación y proteínas estudiadas en conjunto, se ven moduladas en respuesta a las bajas dosis de radiación ionizante en las células GM14511. La integración de múltiples datos y la construcción de redes funcionales de genes pueden proporcionar nuevos datos sobre los mecanismos moleculares que están operando. Para ello, se valorarán las metodologías descritas por Cabezas-Wallscheid N et al (Identification of regulatory networks in HSCs and their immediate progeny via integrated proteome, transcriptome, and DNA methylome analysis.Cell Stem Cell. 2014 Oct 2;15(4):507-22. doi: 10.1016/j.stem.2014.07.005. Epub 2014 Aug 21), Pineda S et al (Framework for the Integration of Genomics, Epigenomics and Transcriptomics in Complex Diseases. Hum Hered. 2015;79(3-4):124-36. doi: 10.1159/000381184. Epub 2015 Jul 28) y Tyekucheva S et al (Integrating diverse genomic data using gene sets. Genome Biol. 2011; 12(10): R105. Epub 2011/10/25. doi: 10.1186/gb-2011-12-10-r105).
También, se estudiarán las redes biológicas y las vías de señalización de las regiones identificadas en los análisis individuales utilizando entre otras las herramientas bioinformáticas «Ingenuity Pathway Analysis, DAVID» (https://david.ncifcrf.gov/) y STRING (http://string-db.org/).
d) Hitos de la tarea 2
– H2.1: Almacenamiento de datos (Mes 18) (UAM).
– H2.2: Análisis bioinformáticos/bioestadísticos individuales. (Mes 24) (UAM).
– H2.3: Análisis integrativos (Mes 33) (UAM).
A.5.4.3 Tarea 3. Identificación de marcadores de sensibilidad individual a la radiación.
a) Sub-tarea 3.1. Selección y recogida de muestras de trabajadores sanitarios expuestos ocupacionalmente a bajas dosis de radiación ionizante (URV).
La selección de los sujetos de estudio se realizará de acuerdo a los puestos de trabajo catalogados como profesionalmente expuestos a radiaciones ionizantes según el Real Decreto 783/2001 de 6 julio, por el que se aprueba el Reglamento sobre protección sanitaria contra radiaciones ionizantes (modificado el 26 de julio de 2002, BOE nº 178). Los sujetos que participarán en este estudio pertenecen todos ellos a un grupo de trabajadores del Hospital Universitario Sant Joan de Reus expuestos a muy bajas dosis de radiación (n = 50). La muestra seleccionada se estratificará posteriormente en diferentes subgrupos atendiendo a la dosis recibida, el sexo y un rango de edades comprendido dentro de la edad laboral. Como grupo control se seleccionarán personas no expuestas a la radiación (n = 50). Previamente, se obtendrá la aprobación del Comité Ético del Hospital Universitario Sant Joan de Reus y del personal seleccionado, el preceptivo consentimiento informado para participar en este estudio.
Se utilizarán linfocitos obtenidos de sangre periférica de los trabajadores sanitarios seleccionados para la validación in vivo de la genes de radiosensibilidad identificados en los estudios in vitro. Estas muestras serán rutinariamente procesadas y crioconservadas, siguiendo procedimientos experimentales ya establecidos.
b) Sub-tarea 3.2. Validación de genes de radiosensibilidad en linfocitos de sangre periférica mediante RT-PCR cuantitativa a tiempo real (UAB).
Una vez analizados mediante herramientas bioinformáticas los resultados del análisis del transcriptoma de las células GM14511 se realizará un filtrado por «fold change». Ello permite seleccionar los genes que cambian más radicalmente su expresión para su posterior aplicación como biomarcadores de exposición en linfocitos de trabajadores sanitarios expuesto a bajas dosis de radiación. Previo a su uso como biomarcadores deben comprobarse los cambios identificados en las micromatrices mediante ensayos de expresión de genes individuales. Esta validación se realiza mediante PCR cuantitativa (qRT-PCR), en la que se emplea para cada gen cDNA (obtenido por retrotranscripción del ARN total) que actúa de molde, un par de cebadores específicos, desoxinucleótidos trifosfato y una enzima ADN polimerasa estable que cataliza la reacción de amplificación del RNA problema (convertido a cDNA). Se añade a todo ello una sustancia marcada con un fluoróforo que, en un termociclador dotado de sensores para medir la fluorescencia, al ser excitado con luz de longitud de onda adecuada, nos permitirá medir la tasa de generación del producto específico. Esta medida se realizará después de cada ciclo de amplificación proporcionando una estimación de la cantidad inicial de RNA problema, que es inversamente proporcional al número de ciclos de PCR necesarios para obtener una cantidad detectable de producto.
Una vez validados en la línea celular CD3+ Pan T los genes candidatos cuya expresión cambia de manera más significativa en células irradiadas, se analizarán estos mismos candidatos en muestras de ARN de linfocitos de sangre periférica de trabajadores sanitarios a fin de detectar diferencias interindividuales.
c) Sub-tarea 3.3. Validación de genes de radiosensibilidad en linfocitos de sangre periférica mediante modificación del ADN con bisulfito y pirosecuenciación (UAM).
Los datos obtenidos del metiloma en células CD3+ Pan T que muestren una variación más relevante en los niveles de metilación del ADN serán posteriormente validados mediante la modificación del ADN con bisulfito y pirosecuenciación. Las marcas epigenéticas validadas serán objeto de un segundo análisis, esta vez, en muestras de linfocitos de sangre periférica de trabajadores sanitarios para determinar si existen variaciones inter-individuales en los patrones de metilación del ADN.
d) Sub-tarea 3.4. Validación de genes de radiosensibilidad en linfocitos de sangre periférica mediante Western blot o SRM (URV).
Después de la identificación de potenciales biomarcadores, los datos obtenidos del proteoma serán posteriormente validados mediante Western Blotting o ELISA y/o mediante la comparación entre los métodos de espectrometría de masas de proteínas y péptidos (SRM i iTRAQ) en células CD3+ Pan T. Las marcas validadas serán objeto de un segundo análisis en muestras de sangre periférica de trabajadores sanitarios.
e) Sub-tarea 3.5 Identificación de polimorfismos funcionales en genes de radiosensibilidad candidatos (UAM).
Una vez analizados los perfiles de expresión transcripcional y proteica, así como los patrones de metilación del ADN en los genes de radiosensibilidad candidatos, procederemos a la pirosecuenciación de dichos genes con el fin de identificar posibles variantes alélicas funcionales. Con ello, se completaría el análisis de los genes de radiosensibilidad a nivel de la propia secuencia nucleotídica.
f) Hitos de la tarea 3.
– H3.1: Selección y estratificación de la población problema (Mes 19) (URV).
– H3.2: Recogida de linfocitos de sangre periférica de la muestra problema (Mes 21) (URV).
– H3.3: Validación funcional de genes de radiosensibilidad en linfocitos de sangre periférica (Mes 32) (UAB, UAM, URV).
– H3.4: Identificación de polimorfismos funcionales en genes de radiosensibilidad (Mes 32) (UAM).
A.5.5 Tablas descriptivas de las distintas tareas:
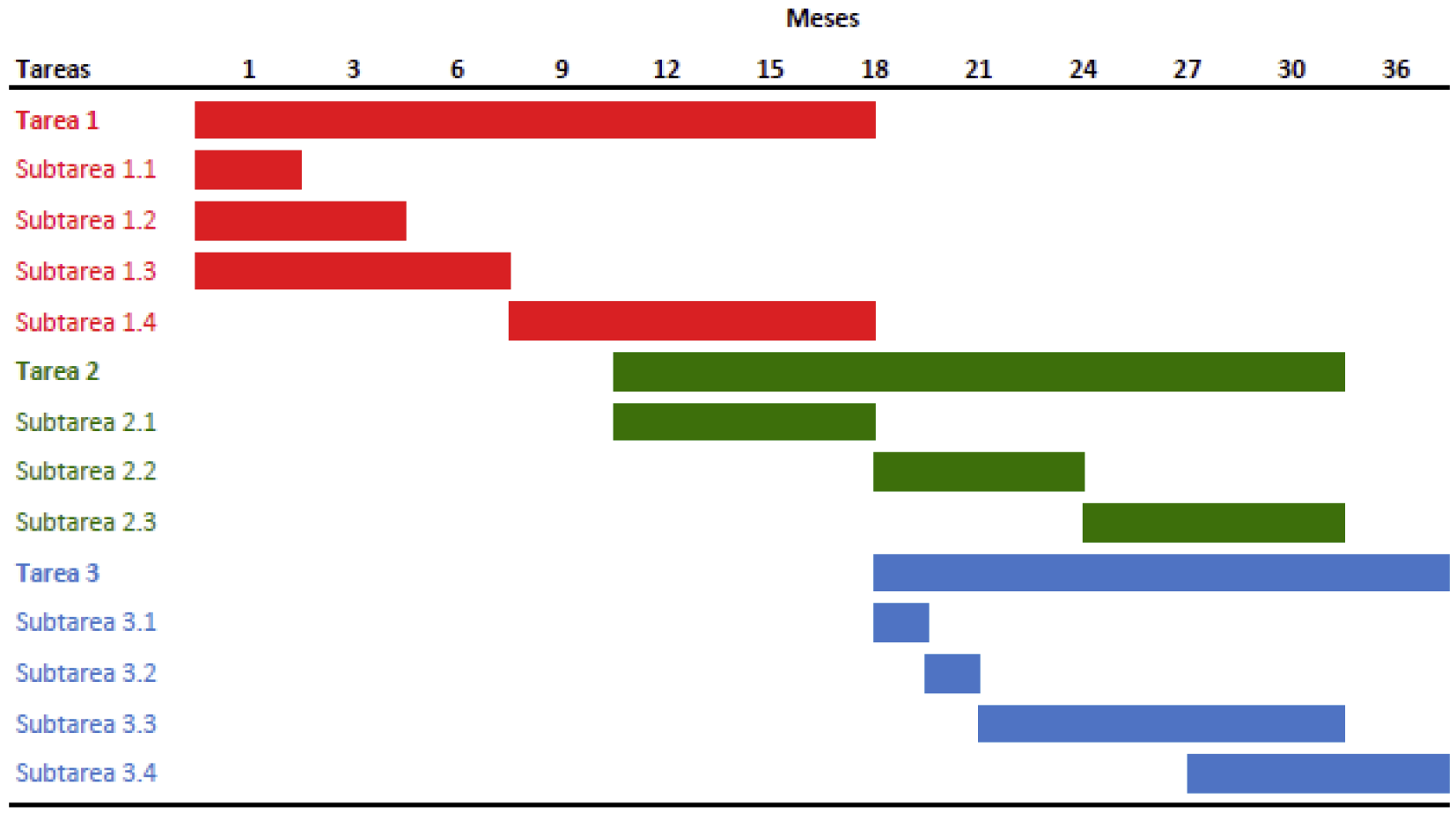
Fecha de inicio de la actividad: Mes 1. Fecha de finalización de la actividad: Mes 18.
Tarea 1: Generación de datos ómicos en células linfoides CD3+ Pan T irradiadas.
Objetivos:
• Irradiar células CD3+ Pan T, derivadas de un único vial, a dos dosis de rayos X.
• Obtener tres replicados biológicos a las 4h post-irradiación de cada situación experimental.
• Llevar a cabo análisis ómicos en los tres replicados a los distintos tiempos para cada situación experimental.
• Generar los datos ómicos para su análisis bioinformático (tarea 2).
Descripción:
• Sub-tarea 1.1. Irradiación de células CD3+ Pan T a 0,1 Gy y 1 Gy (UAM).
• Sub-tarea 1.2. Análisis del transcriptoma (UAB).
• Sub-tarea 1.3. Análisis epigenómico de los patrones de metilación del ADN y modificación de histonas (UAM).
• Sub-tarea 1.4. Análisis del proteoma y fosfo-proteoma (URV).
Resultados previstos:
R1. Datos ómicos para cada dosis y tiempo en cada situación experimental (Mes 18).
Fecha de inicio de la actividad: Mes 12. Fecha de finalización de la actividad: Mes 33.
Tarea 2: Análisis bioinformático/bioestadístico de los conjuntos de datos ómicos obtenidos de células CD3+ Pan T irradiadas.
Objetivos:
• Almacenar de manera segura los datos ómicos.
• Realizar un análisis bioinformático/bioestadístico de manera individual de cada conjunto de datos ómicos.
• Llevar a cabo un análisis bioinformático/bioestadístico de manera colectiva de todos los conjuntos de datos ómicos.
• Integrar los conjuntos de datos ómicos en redes y rutas de señalización.
Descripción:
• Sub-tarea 2.1. Almacenaje de los distintos datos ómicos (UAM).
• Sub-tarea 2.2. Análisis bioinformático/bioestadístico datos ómicos (UAM).
• Sub-tarea 2.3. Análisis bioinformático/bioestadístico integrado (UAM).
Resultados previstos:
R2. Integración de datos ómicos mostrando redes y rutas de señalización relevantes alteradas en respuesta a las bajas dosis de radiación en células linfoides (Mes 33).
Fecha de inicio de la actividad: Mes 18. Fecha de finalización de la actividad: Mes 36.
Tarea 3: Identificación de marcadores de sensibilidad individual a la radiación.
Objetivos:
• Identificar genes de radiosensibilidad individual mediante un análisis in vivo de linfocitos de sangre periférica obtenidos a partir de una muestra de trabajadores sanitarios expuestos ocupacionalmente a bajas dosis de radiación.
Descripción:
• Sub-tarea 31. Selección y recogida de muestras de trabajadores sanitarios expuestos ocupacionalmente a bajas dosis de radiación ionizante (URV).
• Sub-tarea 3.2. Análisis transcripcional de genes de radiosensibilidad en linfocitos de sangre periférica (UAB).
• Sub-tarea 3.3. Estudio de los patrones de metilación del ADN de genes de radiosensibilidad en linfocitos de sangre periférica (UAM).
• Sub-tarea 3.4. Análisis de expresión proteica de radiosensibilidad en linfocitos de sangre periférica (URV).
Resultados previstos.
R1. Identificación de marcadores genéticos de radiosensibilidad individual en células linfoides humanas (Mes 36).
A.5.6 Cronograma del plan de trabajo.
A.6 Descripción de los grupos participantes.
A.6.1 Participante n.º 1.
Grupo de Genética y Biología Molecular del Cáncer (GGBMC), Dpto de Biología Celular e Inmunología, Centro de Biología Molecular Severo Ochoa-Universidad Autónoma de Madrid (CBMSO-UAM).
Descripción del grupo participante:
El Centro de Biología Molecular «Severo Ochoa» (CBMSO) fue fundado en 1975 como un centro mixto entre la Universidad Autónoma de Madrid (UAM) y el Consejo Superior de Investigaciones Científicas (CSIC). En él trabajan varios de los grupos más activos en el campo de la genética y la biología molecular en España. El CBMSO también juega un papel importante en la formación de jóvenes investigadores. En los últimos 15 años el GGBMC ha generado varios modelos de ratón para identificar genes supresores de tumores y genes de resistencia/ susceptibilidad al cáncer implicados en el desarrollo de los linfomas tímicos inducidos por los rayos gamma (gRITL) en el marco de proyectos nacionales e internacionales. El uso de híbridos F1 entre cepas de ratón sensibles a los gRITLs (RF, C57BL/6, BALB/c) nos permitió demostrar la participación, entre otros, de p15, p16, p73 y Pten, que se inactivan por deleciones y/o los cambios epigenéticos. También demostramos, en colaboración con el Dr. M Esteller (CNIO), que Cdh1 y Cdh2 se inactivan epigenéticamente en las células del estroma del timo. Recientemente, hemos identificado, en colaboración con el Dr. M Malumbres, (CNIO), 41 microRNAs con expresión reducida en los gRITL. Uno de ellos (miR203) controla la expresión de dos oncogenes importantes (Abl y Bcr) en el desarrollo de los linfomas. Utilizando cepas de ratones consómicos y congénicos interespecíficos generadas entre la cepa resistente a gRITL SEG/Pas y la sensible C57BL/6, por los Dres. JL Guenet y X Montagutelli (Instituto Pasteur) en el marco de un proyecto colaborativo, se demostró que el gen Anxa1 juega un papel importante en la inducción de la resistencia a los gRITLs en las células estromáticas del timo. En el marco de un proyecto financiado por el Consejo de Seguridad Nuclear, hemos estudiado la respuesta radioadaptativa en un modelo in vivo de ratón demostrando un efecto genético diferencial de dicha respuesta en ambos sexos en el que está implicado, entre otros, un gen supresor de tumores muy importante como es Trp53.
Papel en el proyecto del grupo participante:
El grupo de la UAM contribuirá al proyecto como coordinador científico. Experimentalmente nos implicaremos en la sub-tarea 1.1, donde realizaremos los cultivos celulares y procederemos a la irradiación de las células para su distribución entre los participantes para su análisis ómico. La UAM será responsable de los análisis del epigenoma en células GM14511 irradiadas y en muestras obtenidas de los trabajadores expuestos ocupacionalmente a la radiación (sub-tarea 1.2 y sub-tarea 3.3)
Miembros del grupo participantes en el proyecto:
Javier Santos (Prof. titular Genética) será el IP del grupo UAM. Ha participado activamente en proyectos de I + D financiados por EURATOM. Es el representante de la UAM en MELODI y también participa como IP del equipo en un proyecto financiado por Consejo de Seguridad Nuclear (CSN). Los estudios de metilación se llevarán a cabo bajo la supervisión de José Fernández-Piqueras (Catedrático Genética), director del laboratorio y líder del grupo de Genética del Cáncer en el CBMSO. Tiene experiencia como IP en numerosos proyectos financiados por el Plan Nacional, cuyos objetivos son el estudio de los mecanismos genéticos y epigenéticos responsables de la inducción de los gRITLs. Pilar López-Nieva (PhD senior) colaborará en los estudios de metilación del ADN. Ella tiene experiencia en este campo ya que trabajó durante más de cinco años en uno de los grupos epigenéticos líderes en el mundo bajo la dirección del Dr. Esteller.
La Dra. Iria González Vasconcellos con amplia experiencia en el análisis epigenéticod e histonas realizará el estudio de las modificaciones de histons propuestas.
Los análisis informáticos contarán con el asesoramiento del Dr, Pablo Fernández Navarro (CIBERESP), que cuenta con experiencia tanto el campo de la Epidemiología Ambiental (estudios geográficos y de mortalidad por cáncer y enfermedades cardio-respiratatorias utilizando registros individuales y poblacionales) como en el de la Epidemiología Genética (Identificación de genes de susceptibilidad en cáncer de mama, en linfomas linfoblásticos T humanos y de ratón inducidos por radiación gamma, densidad mamográfica y de resistencia a drogas en trastornos psiquiatricos mayores a partir de datos genómicos).
Publicaciones recientes relevantes para el proyecto:
– López-Nieva et al (2015) Transcriptomic analysis reveals sex-specific differences in the expression of Dcl1 and Fis1 genes in the radio-adaptive response of thymocytes to Trp53-mediated apoptosis. BMC Genomics. Submitted: GICS-D-15-01148.
– Roncero e t al (2015). Contribution of JAK2 mutations to T-cell lymphoblastic lymphoma development. Leukemia doi 10.1038/leu.2015.202.
– González-Gugel et al (2013). Down-regulation of specific miRNAs enhances expression of the gene Smoothened and contributes to T-cell lymphoblastic lymphoma development. Carcinogenesis 34: 902-8.
– López-Nieva et al (2012). EPHA7, a new target gene for 6q deletion in T-cell lymphoblastic lymphomas. Carcinogenesis 33: 452-8.
– Bueno et al. (2011).Combinatorial effects of microRNAs to suppress the Myc oncogenic pathway. Blood 117: 6255-66.
– Santos et al (2010). The stromal gene encoding the CD274 antigen as a genetic modifier controlling survival of mice with gamma-radiation-induced T-cell lymphoblastic lymphomas. Oncogene 29: 5265-73.
– Santos et al (2009). A role for stroma-derived annexin A1 as mediator in the control of genetic susceptibility to T-cell lymphoblastic malignancies through prostaglandin E2 secretion. Cancer Res 69: 2577-87.
– Fernandez-Navarro P, González-Neira A, Pita G, Díaz-Uriarte R, Tais Moreno L, Ederra M, Pedraz-Pingarrón C, Sánchez-Contador C, Vázquez-Carrete JA, Moreo P, Vidal C, Salas-Trejo D, Stone J, Southey MC, Hopper JL, Pérez-Gómez B, Benitez J, Pollan M. Genome wide association study identifies a novel putative mammographic density locus at 1q12-q21. Int J Cancer. 2015 May 15;136(10):2427-36. doi: 10.1002/ijc.29299.
– Lindström S, Thompson DJ, Paterson AD, Li J, Gierach GL, Scott C, Stone J, Douglas JA, dos-Santos-Silva I, Fernandez-Navarro P, Verghase J, Smith P, Brown J, Luben R, Wareham NJ, Loos RJ, Heit JA, Pankratz VS, Norman A, Goode EL, Cunningham JM, deAndrade M, Vierkant RA, Czene K, Fasching PA, Baglietto L, Southey MC, Giles GG, Shah KP, Chan HP, Helvie MA, Beck AH, Knoblauch NW, Hazra A, Hunter DJ, Kraft P, Pollan M, Figueroa JD, Couch FJ, Hopper JL, Hall P, Easton DF, Boyd NF, Vachon CM, Tamimi RM. Genome-wide association study identifies multiple loci associated with both mammographic density and breast cancer risk. Nat Commun. 2014 Oct 24;5:5303.
– Li J, Foo JN, Schoof N, Varghese JS, Fernandez-Navarro P, Gierach GL, Quek ST, Hartman M, Nord S, Kristensen VN, Pollán M, Figueroa JD, Thompson DJ, Li Y, Khor CC, Humphreys K, Liu J, Czene K, Hall P. Large-scale genotyping identifies a new locus at 22q13.2 associated with female breast size. J Med Genet. 2013 Oct;50(10):666-73.
A.6.2 Participante n.º 2.
Grupo de Integridad Genómica (GIG), Departamento de Biología Cellular, Fisiología e Inmunología. Universidad Autónoma de Barcelona (UAB).
Descripción del grupo participante:
El equipo de investigación Genome Integrity de la UAB (GIG) ha publicado en revistas científicas de 1er decil i 1er cuartil en las áreas de Genética y Herencia, Toxicología, Oncología y Gerontología y Geriatría. Los resultados de este equipo han contribuido a la comprensión de los mecanismos de inestabilidad genómica dependiente de disfunción telomérica, que se reconoce como un importante factor promotor de la tumorigénesis en humanos. Los estudios llevados a cabo por este equipo de investigación demuestran que el comportamiento mitótico de los puentes de cromatina originados por la fusión de cromosomas con telómeros erosionados determina los distintos tipos de anomalías generadas en células premalignas. Estudios llevados a cabo en este laboratorio demuestran que los puentes que resisten la tensión del huso mitótico sin romperse originan fenómenos de tetraploidización y segregación anómala de los cromosomas. Por su parte, la rotura de los puentes origina translocaciones desequilibradas y amplificación génica. La presencia de telómeros excesivamente cortos en las células provoca además una mayor radiosensibilidad, que puede contribuir al aumento de la radiosensibilidad que se observa con la edad. En un estudio reciente, el equipo de investigación demuestra que las dosis recibidas por la mama tras una sola exploración mamográfica inducen un aumento significativo en la frecuencia de roturas de doble cadena de DNA en las células epiteliales mamarias envejecidas, pero no en las jóvenes. Ello se debe a que las células envejecidas poseen vías de respuesta al daño en el DNA menos eficaces que las jóvenes. Estudios previos de este equipo de investigación demuestran también la importancia de una detección rápida y completa de las lesiones presentes para mantener la integridad del genoma. Tanto las anomalías en la cinética de respuesta al daño en el DNA como la detección incompleta de las lesiones se traduce en una elevada radiosensibilidad e inestabilidad del genoma de las células. Además, este equipo de investigación ha contribuido a comprender la importancia del ambiente en el que se producen las lesiones: las lesiones que han quedado secuestradas en los micronúcleos son incapaces de reclutar de manera eficiente las proteínas necesarias para su reparación. Los micronúcleos, que se habían considerado hasta hace poco indicadores de inestabilidad genómica, emergen ahora como una fuente de inestabilidad al mismo tiempo.
Papel en el proyecto del grupo participante:
El grupo de la UAB será responsable de la sub-tarea 1.2, donde realizaremos el análisis del transcriptoma del modelo experimental. La UAB participará además en la validación de los candidatos identificados y en los análisis del transcriptoma en las muestras obtenidas de los trabajadores expuestos ocupacionalmente a la radiación (sub-tarea 3.2).
Miembros del grupo participantes en el proyecto:
Anna Genescà (Catedrática de área Biología Celular) será el IP del grupo UAB. Ha participado activamente en proyectos de I + D financiados por EURATOM, habiendo sido work package leader o investigador principal en un total de 6 proyectos europeos en el 3º, 4º, 5º, 6º y 7º Programa Marco. En cuanto a la financiación nacional, ha sido investigadora principal de cinco proyectos de investigación del Consejo de Seguridad Nuclear y de tres proyectos del Programa Acciones Especiales del Ministerio de Educación y Ciencia y similares. Es el representante de la UAB en MELODI. Mariona Terradas (investigadora posdoctoral) y Marta Martín (Profesora Titular Interina del área Biología Celular) participarán en los estudios de transcriptoma y los estudios funcionales de los nuevos genes relevantes en la respuesta a bajas dosis de radiación. Tienen experiencia en este campo ya que han participado en el proyecto Dark Risk: Studies on a cohort of Serbian children exposed to x-irradiation to determine the contribution of the non-coding genome to susceptibility at low doses.
Publicaciones más relevantes para el Proyecto:
– Domínguez D, Feijoo P, Bernal A, Ercilla A, Agell N, Genescà A, Tusell L. Centrosome aberrations in human mammary epithelial cells driven by cooperative interactions between p16INK4a deficiency and telomere-dependent genotoxic stress. Oncotarget. 2015 Sep 29;6(29):28238-56.
– Russo A, Pacchierotti F, Cimini D, Ganem NJ, Genescà A, Natarajan AT, Pavanello S, Valle G, Degrassi F. Genomic instability: Crossing pathways at the origin of structural and numerical chromosome changes. Environ Mol Mutagen. 2015 Aug;56(7):563-80.
– Hernández L, Terradas M, Camps J, Martín M, Tusell L, Genescà A. Aging and radiation: bad companions. Aging Cell. 2015 Apr;14(2):153-61.
– Martín M, Terradas M, Hernández L, Genescà A. γH2AX foci on apparently intact mitotic chromosomes: not signatures of misrejoining events but signals of unresolved DNA damage. Cell Cycle. 2014;13(19):3026-36.
– Feijoo P, Dominguez D, Tusell L, Genesca A. Telomere-dependent genomic integrity: evolution of the fusion-bridge-breakage cycle concept. Curr Pharm Des. 2014;20(41):6375-85.
– Hernández L, Terradas M, Martín M, Tusell L, Genescà A. Highly sensitive automated method for DNA damage assessment: gamma-H2AX foci counting and cell cycle sorting. Int J Mol Sci. 2013 Jul 30;14(8):15810-26.
– Hernández L, Terradas M, Martín M, Feijoo P, Soler D, Tusell L, Genescà A. Increased mammogram-induced DNA damage in mammary epithelial cells aged in vitro. PLoS One. 2013 May 7;8(5):e63052.
– Terradas M, Martín M, Hernández L, Tusell L, Genescà A. Is DNA damage response ready for action anywhere? Int J Mol Sci. 2012;13(9):11569-83.
– Frias C, Pampalona J, Genesca A, Tusell L. Telomere dysfunction and genome instability. Front Biosci (Landmark Ed). 2012 Jun 1;17:2181-96.
– Pampalona J, Frías C, Genescà A, Tusell L. Progressive telomere dysfunction causes cytokinesis failure and leads to the accumulation of polyploid cells. PLoS Genet. 2012;8(4):e1002679.
– Martín M, Terradas M, Tusell L, Genescà A. ATM and DNA-PKcs make a complementary couple in DNA double strand break repair. Mutat Res. 2012; 751:29-35.
– Terradas M, Martín M, Hernández L, Tusell L, Genescà A. Nuclear envelope defects impede a proper response to micronuclear DNA lesions. Mutat Res. 2012 Jan 3;729(1-2):35-40.
– Genescà A, Pampalona J, Frías C, Domínguez D, Tusell L. Role of telomere dysfunction in genetic intratumor diversity. Adv Cancer Res. 2011;112:11-41.
– Tusell L, Pampalona J, Soler D, Frías C, Genescà A. Different outcomes of telomere-dependent anaphase bridges. Biochem Soc Trans. 2010 Dec;38(6):1698-703.
– Terradas M, Martín M, Tusell L, Genescà A. Genetic activities in micronuclei: is the DNA entrapped in micronuclei lost for the cell? Mutat Res. 2010 Jul-Sep;705(1):60-7.
– Pampalona J, Soler D, Genescà A, Tusell L. Whole chromosome loss is promoted by telomere dysfunction in primary cells. Genes Chromosomes Cancer. 2010 Apr;49(4):368-78.
– Pampalona J, Soler D, Genescà A, Tusell L. Telomere dysfunction and chromosome structure modulate the contribution of individual chromosomes in abnormal nuclear morphologies. Mutat Res. 2010 Jan 5;683(1-2):16-22.
A.6.3 Participante n.º 3.
Grupo de Toxicología y Salud Mediambiental (TecnATox), Unidad de Investigación de Lípidos y Arteriosclerosis (URLA) de la facultad de Medicina y Ciencias de la Salud, y el Grupo de investigación de oncología radioterápica (GIOR) del Hospital Universitario Sant Joan de Reus, Universidad Rovira i Virgili (URV).
El grupo de la URV participará en este proyecto con científicos TecnATox y URLA. TecnATox, el laboratorio de Toxicología y Salud Mediambiental fue uno de los primeros grupos de Europa en abordar el risk assessment en salud humana, derivado de la exposición a agentes ambientales. Durante más de 30 años, se han llevado a cabo diferentes estudios para valorar los efectos sistémicos debido a bajas dosis de radiación y toxicos mediambientales. Además se estan llevando a cabo diversos estudios en los que se relacionan diferentes patologías con los efectos físicos y químicos de las radiaciones tanto externas como internas. Estos grupos han colaborado en el estudio del impacto de Chernobil en Cataluña. URV ha participado en diversos proyectos de EURATOM, siendo los más recientes CEREBRAD y SOPRANO. Además forma parte de las plataformas MELODI y OPERRA. La URV creó el Centro de Ciéncias Ómicas (COS), centro especializado en técnicas metabolomicas, proteomicas, transcriptomicas, y genómicas.
URV (TecnATox-URLA) tienen experiencia en regulación epigenética y dentro del proyecto será responsable de la sub-tarea 1.4, donde se realizará el análisis del proteoma y fosfo-proteoma de las muestras generadas, de la sub-tarea 3.1, donde se seleccionaran y obtendrán muestras de trabajadores sanitarios expuestos ocupacionalmente a bajas dosis de radiación, subtarea 3.4 donde se validarán los genes radiosensibles de sangre periférica.
Miembros del grupo participantes en el proyecto:
Victoria Linares Vidal (Profesora Agregada de Fisiología) será la IP del grupo URV. Ha llevado a cabo diversos estudios para determinar los mecanismos implicados en los efectos cancerígenos y no cancerígenos debido a la exposición a radiaciones externas y internas. En la actualidad es la representante de la URV en MELODI y OPERRA. Ha participado en proyectos de I + D financiados por EURATOM, habiendo sido work package leader o investigador principal en varios de ellos. Montserrat Bellés es profesora Titular de Fisiología. Sus principales líneas de trabajo son el estudio de los efectos in vivo de radionucleidos a nivel sistémico. Joan Carles Vallvé es un investigador con una amplia experiencia como responsable de grupo y como IP en numerosos proyectos financiados por el Plan Nacional, cuyos objetivos son el estudio de los mecanismos genéticos y epigenéticos a nivel cardiovascular. Tiene una amplia experiencia en las ciencias ómicas.
Publicaciones más relevantes para el Proyecto:
– Serra N, Rosales R, Masana L, Vallvé JC. Simvastatin Increases Fibulin-2 Expression in Human Coronary Artery Smooth Muscle Cells via RhoA/Rho-Kinase Signaling Pathway Inhibition. PLoS One. 2015 Jul 24;10(7):e0133875.
– Ibarretxe D, Girona J, Plana N, Cabré A, Heras M, Ferré R, Merino J, Vallvé JC, Masana L. FABP4 plasma concentrations are determined by acquired metabolic derangements rather than genetic determinants. Nutr Metab Cardiovasc Dis. 2015 Sep;25(9):875-80.
– Guardiola M, Vallvé JC, Zaina S, Ribalta J. [Epigenetics in atherosclerosis]. Clin Investig Arterioscler. 2015 Jun 15. pii: S0214-9168(15)00054-6.
– Echeverria P, Guardiola M, González M, Vallvé JC, Bonjoch A, Puig J, Clotet B, Ribalta J, Negredo E. Association between polymorphisms in genes involved in lipid metabolism and immunological status in chronically HIV-infected patients. Antiviral Res. 2015 Feb;114:48-52.
– Guardiola M, Oliva I, Guillaumet A, Martín-Trujillo Á, Rosales R, Vallvé JC, Sabench F, Del Castillo D, Zaina S, Monk D, Ribalta J. Tissue-specific DNA methylation profiles regulate liver-specific expression of the APOA1/C3/A4/A5 cluster and can be manipulated with demethylating agents on intestinal cells. Atherosclerosis. 2014 Dec;237(2):528-35.
– Vallvé JC, Serra N, Zalba G, Fortuño A, Beloqui O, Ferre R, Ribalta J, Masana L. Two variants in the fibulin2 gene are associated with lower systolic blood pressure and decreased risk of hypertension. PLoS One. 2012;7(8):e43051.
– Guardiola M, Alvaro A, Vallvé JC, Rosales R, Solà R, Girona J, Serra N, Duran P, Esteve E, Masana L, Ribalta J. APOA5 gene expression in the human intestinal tissue and its response to in vitro exposure to fatty acid and fibrate. Nutr Metab Cardiovasc Dis. 2012 Sep;22(9):756-62.
– Alvaro A, Rosales R, Masana L, Vallvé JC. Polyunsaturated fatty acids down-regulate in vitro expression of the key intestinal cholesterol absorption protein NPC1L1: no effect of monounsaturated nor saturated fatty acids. J Nutr Biochem. 2010 Jun;21(6):518-25.
– Horra A, Salazar J, Ferré R, Vallvé JC, Guardiola M, Rosales R, Masana L, Ribalta J. Prox-1 and FOXC2 gene expression in adipose tissue: A potential contributory role of the lymphatic system to familial combined hyperlipidaemia. Atherosclerosis. 2009 Oct;206(2):343-5.
– Alvaro A, Solà R, Rosales R, Ribalta J, Anguera A, Masana L, Vallvé JC. Gene expression analysis of a human enterocyte cell line reveals downregulation of cholesterol biosynthesis in response to short-chain fatty acids. IUBMB Life. 2008 Nov;60(11):757-64.
– Bellés M, Heredia L, Llovet MI, Linares V. Brain development behavioural effects after internal (137)cesium exposure. GD12 vs postnatal. Int J Dev Neurosci. 2015 Dec;47(Pt A):29.
– Bellés M, Heredia L, Llovet I, Linares V. Neurocognitive effects of exposure to low-dose internal radiation and paraquat in developing offspring mice. Int J Dev Neurosci. 2015 Dec;47(Pt A):29.
– Bellés M, Heredia L, Llovet MI, Linares V. Brain development behavioural effects after internal (137)cesium exposure. GD12 vs postnatal. Int J Dev Neurosci. 2015 Dec;47(Pt A):29.
– Bellés M, Heredia L, Llovet I, Linares V. Neurocognitive effects of exposure to low-dose internal radiation and paraquat in developing offspring mice. Int J Dev Neurosci. 2015 Dec;47(Pt A):29.
– Linares V, Bellés M, Domingo JL. Human exposure to PBDE and critical evaluation of health hazards. Arch Toxicol. 2015 Mar;89(3):335-56.
– Heredia L, Bellés M, Llovet MI, Domingo JL, Linares V. Neurobehavioral effects of concurrent exposure to cesium-137 and paraquat during neonatal development in mice. Toxicology. 2015 Mar 2;329:73-9.
– Bellés M, Linares V, Perelló G, Domingo JL. Human dietary exposure to uranium in catalonia, Spain. Biol Trace Elem Res. 2013 Apr;152(1):1-8.
– Arenas M, Montero Á, de Las Peñas MD, Algara M. The position and current status of radiation therapy after primary systemic therapy in breast cancer: a national survey-based expert consensus statement. Clin Transl Oncol. 2015 Sep 14. [Epub ahead of print]
– Arenas M, Sabater S, Hernández V, Rovirosa A, Lara PC, Biete A, Panés J. Anti-inflammatory effects of low-dose radiotherapy. Indications, dose, and radiobiological mechanisms involved. Strahlenther Onkol. 2012 Nov;188(11):975-81.
– Arenas M, Gil F, Gironella M, Hernández V, Biete A, Piqué JM, Panés J. Time course of anti-inflammatory effect of low-dose radiotherapy: correlation with TGF-beta(1) expression. Radiother Oncol. 2008 Mar;86(3):399-406. Epub 2007 Nov 26.
– Arenas M, Gascón M, Rovirosa À, Hernández V, Riu F, López I, Montero A, Sabater S. The effect of lymphadenectomy and radiotherapy on recurrence and survival in endometrial carcinoma. Experience in a population reference centre. Rep Pract Oncol Radiother. 2014 Oct 14;20(1):50-6.
A.7 Beneficios del proyecto, difusión y explotación de los resultados.
A.7.1 Beneficios esperables del desarrollo del proyecto.
La constitución genética de un individuo es un factor que puede modificar el riesgo sobre la salud de las exposiciones a las bajas dosis de radiación y siguen a la espera de que se identifiquen con exactitud los mecanismos moleculares que están detrás de la susceptibilidad genética a la radiación. Las tecnologías ómicas han aportado mucha información a nivel genético sobre la respuesta celular a las exposiciones con dosis bajas de radiación. Sin embargo, este tipo de análisis ha puesto de manifiesto una gran variabilidad en dicha respuesta dependiendo de cuál haya sido el tipo celular examinado. Por tanto, un análisis centrado en un único tipo celular que integre los datos generados por dichas tecnologías a nivel del ADN, ARN y proteínas resulta una aproximación experimental idónea que puede contribuir a un mejor conocimiento de las bases genéticas de la radiosensibilidad. Nuestro proyecto propone por primera vez llevar a cabo un análisis genético a gran escala que integre los datos obtenidos del análisis transcriptómico, proteómico y epigenómico en un único tipo celular de origen linfoide. Los resultados que se obtengan en este proyecto nos darán una visión integradora de la respuesta de las células linfoides a las bajas dosis de radiación.
Hoy en día, las diferencias individuales en el riesgo sobre la salud de las exposiciones a las bajas dosis de radiación no están contempladas en los sistemas actuales de protección radiológica. Esto solo puede cambiar cuando se conozcan mejor los mecanismos responsables de la radiosensibilidad individual y se puedan disponer de marcadores genéticos que permitan evaluar de manera fiable en cada individuo el riesgo sobre la salud de las bajas dosis de radiación. Este proyecto pretende dar un primer paso hacia la incorporación de marcadores genéticos como biodosímetros en la evaluación del riesgo de los trabajadores expuestos ocupacionalmente a las bajas dosis de radiación, y la inclusión de la radiosensibilidad individual en los programas de protección radiológica.
A.7.2 Difusión y explotación de los resultados.
El nivel de nuestras publicaciones internacionales sería nuestro mejor aval y nuestra incorporación a MELODI y al proyecto Europeo SOPRANO garantizan foros adecuados de discusión y difusión de los resultados.
A.8 Contribución del proyecto al plan estratégico de I+D del Consejo de Seguridad Nuclear (CSN).
El presente proyecto se ajusta perfectamente a las actividades de promoción de la investigación del CSN en el campo la protección radiológica, contempladas en sus planes estratégicos de I+D. El retorno fundamental de las actividades de I+D del CSN es la obtención de conocimiento que contribuya al mejor desarrollo de las labores de evaluación e inspección que dicho organismo tiene asignadas. En este sentido, nuestra propuesta aportaría al CSN nuevos conocimientos sobre los efectos genéticos de las exposiciones a las bajas dosis de radiación, así como información de primera mano sobre la existencia de marcadores genéticos de radiosensibilidad. La identificación de biomarcadores genéticos de radiosensibilidad es un hecho de gran relevancia por cuanto ayudaría a potenciar los protocolos de protección radiológica que actualmente se vienen aplicando. Por tanto, con una mínima sinergia el CSN tendría un importante retorno de las actividades desarrolladas por el proyecto.
Por otro lado, las instituciones participantes en el proyecto participamos de manera activas en las iniciativas promovidas por la Comisión Europea en el marco del programa EURATOM a través de proyectos de investigación competitivos (DARK RISC y SOPRANO). Este sería otro retorno importante para el CSN al promover programas de investigación a nivel nacional complementarios a los de la Comisión Europea. La idea de una cofinanciación de programas europeas en el campo de las bajas dosis de radiación es uno de los pilares en los se apoya el programa Horizonte 2020 en el campo de la protección radiológica. Dicho programa establece la necesidad de promover y mantener programas de investigación en radioprotección. Por lo tanto la sinergia establecida entre nuestro proyecto y el CSN como organismo financiador daría cumplimiento a esta necesidad.
B.1 Instalaciones y equipamiento para la realización del proyecto.
Como personal del CBMSO y de la UAM se tiene acceso a todos los servicios de ambos organismos, además de los del Parque Científico de Madrid, situados todos en el campus universitario de Cantoblanco, necesarios para el desarrollo de este proyecto: tecnologías ómicas y de secuenciación automáticas, cuartos de cultivo celulares adecuadamente equipados e instalaciones radiológicas que incorporan entre su equipamiento un irradiador de rayos X. Además el laboratorio del grupo GGBMC del CBMSO-UAM cuenta con toda la infraestructura necesaria para acometer con garantías el proyecto propuesto: equipos de centrifugación, termocicladores convencionales y en tiempo real, congeladores y ultracongeladores, cabinas de flujo equipo de electroforesis convencionales y en campo pulsado, espectrofotómetros, transiluminadores, sistemas de análisis de da imagen, incubadores de CO2, etc. Por todo ello, no se solicita ayuda para instalaciones o equipos.
En relación con el sistema de almacenamiento y cómputo necesario para llevar a cabo las tareas de bioinformática y análisis estadístico, el grupo UAM cuenta con un Centro de Procesamiento de Datos (CPD), cuyos equipos se encuentran situados en el campus de la UAM. Este sistema que ya da cobertura a otros proyectos del grupo, necesitaría ser renovado en algún punto clave como es el del almacenamiento y así abordar con garantías los objetivos del proyecto. Por ello se solicita hardware que a) vendría a sustituir el actual: como es el servidor general de transferencia de datos y b) alguna unidad adicional de discos duros para la realización de backups de todo el trabajo que se realice.
En las instalaciones de la UAB, el equipo de investigación del proyecto dispone del equipamiento necesario para el desarrollo del proyecto de investigación. Disponemos de una campana de seguridad de flujo vertical para la manipulación de muestras y células humanas, un incubador de CO2, un microscopio invertido para el control del crecimiento de las células en cultivo, un termociclador de gradiente, cubetas de electroforesis, fuentes de alimentación para electroforesis, baños termostáticos, centrífugas refrigeradas y varios microscopios de epifluorescencia equipados con cámaras y sistema para la captura de imágenes. Además, el equipo de investigación va a tener acceso a los servicios core de la Universidad como el servicio de citometría y el de microscopía.
El grupo TecnATox-URLA de la URV tiene acceso a todas las instalaciones de la Universidad así como los servicios técnicos de la misma. Disponemos de toda la infraestructura necesaria para llevar a cabo el proyecto: congeladores/ultracongeladores, incubadores de CO2, espectrofotómetros, transiluminadores, campana de seguridad de flujo vertical, termocicladores, cubetas de electroforesis, fuentes de alimentación, baños termostáticos y centrífugas refrigeradas, microscopios, cromatógrafos, etc. Para la utilización de todo este equipamiento no solicitamos financiación en el proyecto.
B.2 Presupuesto.
El coste total del Acuerdo Específico se ha calculado sobre la base de los costes que se detallan a continuación. Todas las cantidades incluyen todos los impuestos aplicables.
B.2.1 Costes de ejecución.
|
Costes de ejecución. Participante n.º 1- UAM |
Contribución UAM |
Contribución CSN |
|
|---|---|---|---|
|
1. Costes de personal. |
|||
|
1.1 Personal propio UAM. |
|||
|
– Prof. Titular (coste/hora 31,53) x 3 años. |
44.930 |
||
|
– Catedrático (coste/hora 42,27) x 3 años. |
54.951 |
||
|
– Prof. Ayudante Dr (coste /hora 16,17) x 3 años. |
34.565 |
||
|
– Investigador Dr (coste /hora 20,25) x 3 años. |
34.263 |
||
|
– Investigador Dr (coste /hora 20,25) x 3 años. |
34.263 |
||
|
202.972 |
|||
|
1.2 Contratación de un titulado universitario en informática. |
36.000 |
||
|
2. Otros costes directos (servicios, fungible, gastos de viaje). |
|||
|
2.1 Servicios de Genómica para análisis del metiloma. |
37.500 |
||
|
2.2 Material fungible (Kits de extracción de ADN; kits de modificación del ADN con bisulfito; oligos para secuenciación; consumibles informáticos). |
39.550 |
||
|
2.3 Gastos de viaje (reuniones para la gestión del proyecto). |
2.500 |
||
|
Total Costes directos. |
202.972 |
115.550 |
|
|
3. Impuestos (IVA 21%). |
42.624 |
24.266 |
|
|
Costes totales. |
245.596 |
139.816 |
|
|
Porcentaje de contribución al proyecto (%). |
63,7% |
36,3% |
|
|
Costes de ejecución. Participante nº 2- UAB |
Contribución UAB |
Contribución CSN |
|
|---|---|---|---|
|
1. Costes de personal. |
|||
|
1.1 Personal propio UAB. |
|||
|
– Catedrática (coste/hora 50,06) x 3 años. |
100.120 |
||
|
– Prof. Titular Interina (coste/hora 29,35) x 3 años |
19.958 |
||
|
– Técnico de apoyo invest. (coste/hora 24,09) x 3 años |
30.042 |
||
|
150.120 |
|||
|
1.2 Técnico de investigación (12 meses). |
9.010 |
||
|
1.3 Becario predoctoral (6 meses). |
9.506 |
||
|
1.4 Doctor con experiencia acreditada en estudios de transcriptoma. |
12.000 |
||
|
2. Otros costes directos (servicios, fungible, gastos de viaje). |
|||
|
2.1 Arrays análisis de transcriptoma (mRNA y lncRNA). |
12.000 |
||
|
2.2 qRT-PCR análisis individual para validación de genes candidatos. |
9.343 |
||
|
2.3 Servicios centralizados internos y externos. |
4.500 |
||
|
2.4 Gastos de viaje y alojamiento (reuniones para la gestión del proyecto). |
2.350 |
||
|
Total Costes directos. |
150.120 |
58.709 |
|
|
3. Compensación por gastos generales (canon). |
15.605 |
||
|
Total Costes directos + canon. |
150.120 |
74.314 |
|
|
4. Impuestos (IVA 21%). |
15.606 |
||
|
Costes totales. |
150.120 |
89.920 |
|
|
Porcentaje de contribución al proyecto. |
62,60% |
37,40% |
|
|
Costes de ejecución. Participante n.º 3- URV |
Contribución URV |
Contribución CSN |
|
|---|---|---|---|
|
1. Costes de personal. |
|||
|
Profesor agregado, dedicación parcial 3 años (coste hora 36,45). |
73.200 |
||
|
Titular Universidad, dedicación parcial 3 años (coste hora 31,38). |
63.110 |
||
|
Catedrático, dedicación parcial 2 años (coste hora 50,84). |
29.275 |
||
|
Personal técnico, dedicación parcial 3 años (coste hora 24,90) |
26.765 |
||
|
192.350 |
|||
|
Técnico de investigación (dedicación equivalente a 24 meses de trabajo a tiempo completo). |
57.120 |
||
|
2. Otros costes directos (servicios, fungible, gastos de viaje). |
6.000 |
||
|
Total Costes directos. |
192.350 |
63.120 |
|
|
3. Compensación por gastos generales (canon). |
12.624 |
||
|
Total Costes directos + canon. |
192.350 |
75.744 |
|
|
4. Impuestos (IVA 21%). |
15.906 |
||
|
Costes totales. |
192.350 |
91.650 |
|
|
Porcentaje de contribución al proyecto (%). |
67,08% |
32,92% |
|
B.2.2 Distribución del presupuesto aportado por el CSN.
Los costes se distribuirán a lo largo de tres ejercicios presupuestarios, en la forma que se indica en la siguiente tabla:
|
Participante n.º 1- UAM |
1.ª anualidad |
2.ª anualidad |
3.ª anualidad |
Total |
|---|---|---|---|---|
|
Gastos de personal a cargo del proyecto. |
18.000 |
18.000 |
0 |
36.000 |
|
Uso de servicios centralizados de investigación. |
14.000 |
14.000 |
9.500 |
37.500 |
|
Material fungible. |
14.550 |
14.000 |
11.000 |
39.550 |
|
Gastos de viaje. |
1.000 |
1.000 |
500 |
2.500 |
|
Total Costes directos. |
47.550 |
47.000 |
21.000 |
115.550 |
|
Impuestos (21% IVA). |
9.986 |
9.870 |
4.410 |
24.266 |
|
Costes totales a aportar por CSN. |
57.536 |
56.870 |
25.410 |
139.816 |
|
Participante n.º 2- UAB |
1.ª anualidad |
2.ª anualidad |
3.ª anualidad |
Total |
|---|---|---|---|---|
|
Gastos de personal a cargo del proyecto. |
21.506 |
4.505 |
4.505 |
30.516 |
|
Uso de servicios centralizados de investigación. |
1.500 |
1.500 |
1.500 |
4.500 |
|
Material fungible. |
10.000 |
6.000 |
5.343 |
21.343 |
|
Gastos de viaje. |
800 |
800 |
750 |
2.350 |
|
Total Costes directos. |
33.806 |
12.805 |
12.098 |
58.709 |
|
Canon. |
8.986 |
3.404 |
3.215 |
15.605 |
|
Total Costes directos + Canon. |
42.792 |
16.209 |
15.313 |
74.314 |
|
Impuestos (21% IVA). |
8.986 |
3.404 |
3.216 |
15.606 |
|
Costes totales a aportar por CSN. |
51.778 |
19.612 |
18.530 |
89.920 |
|
Participante n.º 3- URV |
1.ª anualidad |
2.ª anualidad |
3.ª anualidad |
Total |
|---|---|---|---|---|
|
Gastos de personal a cargo del proyecto. |
26.100 |
22.000 |
9.000 |
57.120 |
|
Uso de servicios centralizados de investigación. |
2.035 |
1.263 |
702 |
4.000 |
|
Material fungible. |
500 |
400 |
100 |
1.000 |
|
Gastos de viaje. |
400 |
400 |
200 |
1.000 |
|
Costes directos. |
29.035 |
24.083 |
10.002 |
63.120 |
|
Canon. |
5.807 |
4.816 |
2.001 |
12.624 |
|
Total Costes directos + canon. |
34.842 |
28.899 |
12.003 |
75.744 |
|
Impuestos (21% IVA). |
7.317 |
6.069 |
2.521 |
15.906 |
|
Costes totales a aportar por CSN. |
42.159 |
34.968 |
14.523 |
91.650 |
B.2.4 Costes totales del proyecto.
|
Costes de ejecución totales por participante. |
Contribución Institución participante |
Contribución CSN |
|---|---|---|
|
Universidad Autónoma de Madrid. |
245.596 |
139.816 |
|
Universidad Autónoma de Barcelona. |
150.120 |
89.920 |
|
Universidad Rovira i Virgili. |
192.350 |
91.650 |
|
Subtotal. |
588.066 |
321.386 |
|
Totales. |
909.452 |
|
B.2.5 Forma de pago del presupuesto.
El calendario de pagos de este proyecto de I+D es el que se describe a continuación:
• En 2018, un primer pago por importe de 57.536 € a la UAM; de 51.778 € a la UAB; y de 42.159 € a la URV.
• En 2019, un segundo pago por importe de 56.870 € a la UAM; de 19.612 € a la UAB; y de 34.968 € a la URV.
• A la finalización del proyecto, un último pago por importe de 25.410 € a la UAM; de 18.530 € a la UAB; y de 14.523 € a la URV.
Cada uno de los pagos se realizará previa entrega por parte del coordinador del proyecto (UAM) de la documentación mencionada en la Memoria Técnica correspondiente a cada hito y que consistirá en un informe de progreso de las tareas y objetivos marcados en cada período, así como de las facturas emitidas independientemente por cada una de las cuatro instituciones participantes, en las que se reflejan los gastos generados como consecuencia del desarrollo del proyecto en dicho periodo.
En lo referido al último pago (en 2020), se presentará con un mes de antelación un informe que resuma las conclusiones y recomendaciones de la totalidad de los trabajos realizados dentro de este Acuerdo Específico, haciendo referencia a toda la documentación generada a lo largo del mismo. El libramiento del último pago quedará condicionado a la presentación del informe final citado.
Agencia Estatal Boletín Oficial del Estado
Avda. de Manoteras, 54 - 28050 Madrid